細菌べん毛タンパク質輸送の交通整理のしくみを解明
| 原著論文 | Sci Adv 4 (2018) |
|---|---|
| 論文タイトル | Insight into structural remodeling of the FlhA ring responsible for bacterial flagellar type III protein export |
| 研究室サイト | 日本電子YOKOGUSHI協働研究所〈難波 啓一 特任教授(常勤)〉 |
概要
大阪大学大学院生命機能研究科の寺原直矢特任助教(常勤)、井上由美特任技術職員(研究当時。現・京都大学大学院医学研究科教務補佐員)、難波啓一特任教授、南野徹准教授、同大学院理学研究科の今田勝巳教授、金沢大学新学術創成研究機構ナノ生命科学研究所の古寺哲幸教授、安藤敏夫特任教授、名古屋大学大学院理学研究科の内橋貴之教授、九州工業大学大学院情報工学研究院の森本雄祐助教の共同研究グループは、細菌べん毛タンパク質輸送の交通整理のしくみを世界で初めて明らかにしました。
サルモネラ属菌はべん毛と呼ばれる運動器官を使って最適な環境へ移動します。べん毛は約30種類のタンパク質が数分子から数万分子重合してできた超分子複合体です。べん毛は、回転モーターとして働く基部体、ユニバーサルジョイントのフック、らせん型プロペラである繊維の、3つの部分構造で構成されます(図1)。べん毛基部に存在する独自のタンパク質輸送装置は、フックの構築中にはフックタンパク質を輸送します。フックの長さが55nmに到達すると、輸送装置はフックタンパク質の輸送を停止し、繊維形成に関わるタンパク質の輸送を開始します。これまでに、フックの長さを測定するしくみや、フックの完成に伴って起こるタンパク質輸送装置の基質特異性の切り替わるしくみについては詳細に解析されてきました(図2)。しかし、輸送装置が輸送するタンパク質の種類を切り替える、いわば輸送の交通整理のしくみは長い間謎のままでした。
今回、共同研究グループは、高時間高空間分解能で生体分子の観察が可能な高速原子間力顕微鏡を用いて、世界で初めて膜タンパク質であるFlhAの細胞質ドメイン(以降、FlhACと呼ぶ)のリング形成過程をリアルタイムで可視化することに成功しました。FlhAの膜貫通ドメインとFlhACを繋ぐリンカー領域の一部が隣のFlhACに結合することで協働的に9量体リングが形成されること、リンカー領域とFlhACとの相互作用によってフックタンパク質の輸送が停止して繊維タンパク質の輸送が開始することを明らかにしました。本研究成果は、べん毛タンパク質輸送の交通整理のしくみの解明への第一歩とともに、べん毛タンパク質輸送装置と機能的にも構造的にも同じしくみを持つ、病原性細菌のIII型分泌装置のFlhAホモログを直接ターゲットにした創薬スクリーニングが可能になると期待されます。
研究の背景
べん毛は基部体、フック、繊維の、3つの部分構造からなります。べん毛は細胞の内から外に向かって基部体、フック、繊維の順に構築され、べん毛の先端、すなわち細胞から遠い側の端にべん毛構成タンパク質が順序正しく重合することで構築が進みます。べん毛の根元に存在する独自のタンパク質輸送装置がべん毛の構築過程に応じて必要なタンパク質を必要量輸送します。タンパク質輸送装置はフック構築中に時折分子物差しであるFliKを送り出すことで、フックの構築状況をモニターすることができます。フックの長さが55nmになると、FliKがその長さ情報を輸送装置構成タンパク質FlhBに伝え、その結果輸送装置の基質特異性が切り替わることで、フックタンパク質の重合が停止して繊維形成が開始します。しかしながら、輸送装置がどのようにして輸送するタンパク質の種類を変化させるのかは謎のままでした。
本研究の成果
我々は、べん毛タンパク質の輸送に直接関与し、9量体リング構造を作って働く輸送装置タンパク質FlhAに着目し、高速原子間力顕微鏡によりFlhACの動的構造解析を行いました。その結果、FlhACは9量体リングを形成すること、細胞膜貫通ドメインと繋がるリンカー領域が協働的な9量体リング形成に必須であること、9量体リング形成に関わる重要なアミノ酸が異なるアミノ酸に置換されると、9量体リングが形成されなくなるとともに、タンパク質輸送装置の基質特異性がスイッチしないことなどが明らかになりました。以上の結果から、フックが完成すると、FlhAのリンカー領域が隣のFlhAC分子に協働的に結合することでFlhACリングの全体構造が大きく変化し、その結果フックタンパク質の輸送が停止し、繊維形成に関わるタンパク質の輸送が開始することが示唆されました(図3)。
本研究は長い間謎であったタンパク質輸送装置の輸送基質認識モードの切り替えのしくみの可視化に成功するとともに、べん毛タンパク質輸送の交通整理のしくみを明らかにしました。今後は、本研究成果をもとに、フック完成に伴って過渡的にしか存在しないフック完成シグナリング複合体の形成とその機能発現のしくみの解明が期待されます。
本研究は長い間謎であったタンパク質輸送装置の輸送基質認識モードの切り替えのしくみの可視化に成功するとともに、べん毛タンパク質輸送の交通整理のしくみを明らかにしました。今後は、本研究成果をもとに、フック完成に伴って過渡的にしか存在しないフック完成シグナリング複合体の形成とその機能発現のしくみの解明が期待されます。
研究成果のポイント
- べん毛タンパク質輸送装置の構成タンパク質であるFlhAの細胞質ドメインを高速原子間力顕微鏡で解析することにより、FlhAが9量体リングを形成するしくみを解明
- FlhAリングの構造変化によりタンパク質輸送装置の基質特異性が変化することを解明
- 細菌感染症に関わるFlhAホモログを直接ターゲットとした細菌感染症薬のスクリーニングへの応用に期待
本研究成果が社会に与える影響(本研究成果の意義)
抗菌剤は細菌感染症対策の主流ですが、多剤耐性菌の出現により治療困難な感染症が増え、新たな着眼点からの感染症対策が急務となっています。病原細菌が感染する際に用いるIII型分泌装置は細菌特有のもので、しかも病原細菌の生存に必須ではありません。もしIII型分泌装置の働きだけを不活化できる薬剤が見つかれば病原細菌の病原性のみを破壊することが可能となります。III型分泌装置は機能的にも構造的にもべん毛タンパク質輸送装置と似ていますので、本研究成果によりIII型分泌装置の構成タンパク質であるFlhAホモログを直接ターゲットにした創薬スクリーニングが可能となり、社会的な重要課題である新興細菌感染症を制御するための新技術開発に貢献すると期待されます。
特記事項
本研究成果は、2018年4月26日(木)に米国科学誌「Science Advances」(オンライン)に掲載されました。
なお、本研究は、科学研究費補助金、科学技術振興機構(JST)戦略的研究推進事業CRESTおよびさきがけ研究の一環として行われ、大阪大学大学院理学研究科 今田勝巳教授、金沢大学新学術創成研究機構ナノ生命科学研究所 安藤敏夫特任教授と古寺哲幸教授、名古屋大学大学院理学研究科 内橋貴之教授、さらには九州工業大学大学院情報工学研究院 森本雄祐助教の協力を得て行われました。
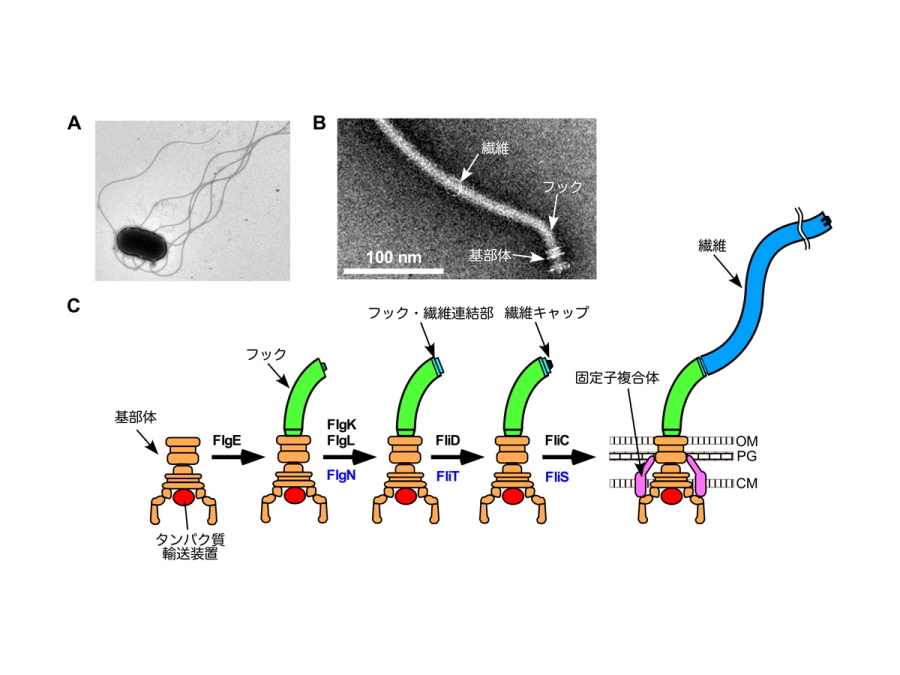
図1.細菌べん毛の構造
(A)サルモネラ属菌の電子顕微鏡写真.(B)サルモネラ属菌から単離精製された電子顕微鏡写真.(C)べん毛の構築過程.OMは外膜、PGはペプチドグリカン層、CMは細胞膜を示す。
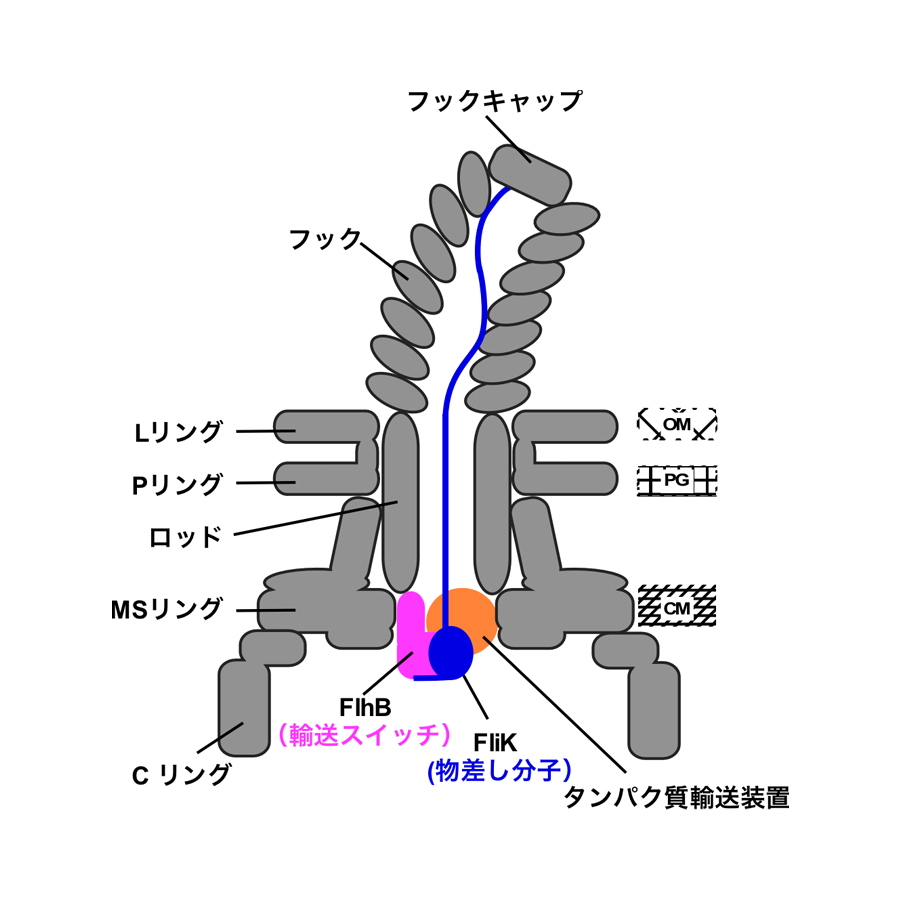
図2.一時的に形成されるフック完成シグナリング複合体の模式図
時折細胞外に送り出されるFliKがフックの長さを測定する分子物差しとして働く。フックの長さが55nmになると、FliKのC末ドメインが輸送スイッチであるFlhBと相互作用し、その結果タンパク質輸送装置の基質特異性が切り替わる。
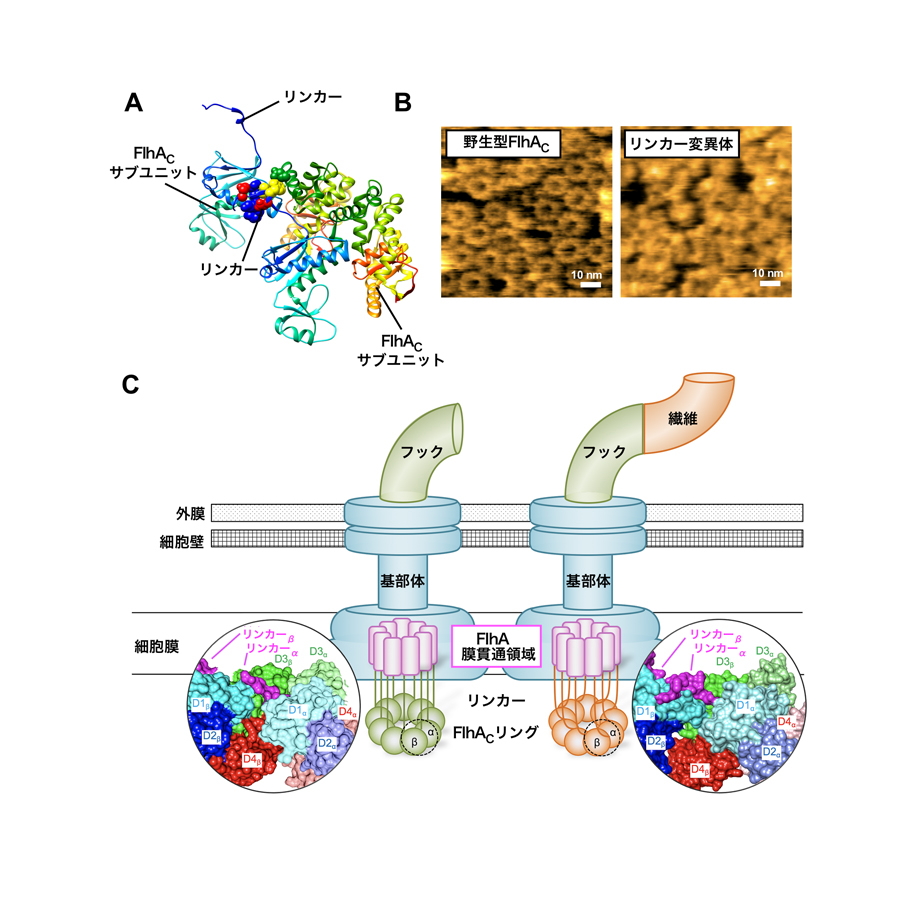
図3.FlhAのリング形成
(A)FlhAの結晶構造.結晶中でリンカー領域が隣のFlhAC分子に結合している様子。(B)FlhACリングの高速原子間力像。リンカーが欠失されると(右)、リング形成が著しく阻害される。(C)FlhACリング構造のリモデリング仮説。FlhAのリンカー領域がFlhACサブユニットに結合すると、FlhACリングの構造が変化し、その結果フックタンパク質の輸送が停止して繊維タンパク質の輸送が開始する。
用語解説
- タンパク質輸送装置
生体膜を透過させてタンパク質を細胞外へ送り出す装置。 - 高速原子間力顕微鏡
カンチレバーと呼ばれる鋭い探針で試料表面をなぞることで試料表面の凹凸を可視化する顕微鏡。金沢大学の安藤敏夫特任教授の研究グループによって高速化および生体試料の観察が実現し、溶液中で生体分子の反応や構造変化の高分解能かつリアルタイムでの観察が可能となった。 - 膜タンパク質
細胞膜などのリン脂質2重層で形成される膜内に存在するタンパク質の一般名称。細胞膜を隔てて各種イオンを流すイオンチャネルや栄養源などの物質を運ぶトランスポーターなどが知られている。
| 原著論文 | Sci Adv 4 (2018) |
|---|---|
| 論文タイトル | Insight into structural remodeling of the FlhA ring responsible for bacterial flagellar type III protein export |
| 著者 | Naoya Terahara (1), Yumi Inoue (1), Noriyuki Kodera (2), Yusuke V. Morimoto (1, 3, 4), Takayuki Uchihashi (2, 5, 6), Katsumi Imada (7), Toshio Ando (2, 8), Keiichi Namba (1, 3), Tohru Minamino (1)
|
| PubMed | 29707633 |
