セントロメアは動くことができる
| 原著論文 | J Cell Biol 216, 101-113 (2017) |
|---|---|
| 論文タイトル | Constitutive centromere-associated network controls centromere drift in vertebrate cells |
| 研究室サイト | 染色体生物学研究室〈深川 竜郎 教授〉 |
概要
セントロメアは、正常な染色体分配にとって必須なゲノム領域である。多くの生物でセントロメアは染色体の特定の場所に1箇所だけ形成されるので、セントロメアの位置は、染色体上に固定されていると考えられていた。深川研究室の堀准教授らは、ニワトリ細胞の培養過程のセントロメアの位置を高精度で解析し、セントロメアが培養の過程で動き得るかどうかを検証した。その結果、長い培養の過程でセントロメアの位置が動くことが明らかになった。しかしながら、短い培養では、セントロメアの位置は、比較的一定であることが判明した。興味深いことに、CENP-UとCENP-Sをノックアウトした細胞では、セントロメアが動きやすいことが判明した。このことから、セントロメアの位置は長い培養過程で動き得るが、通常はCENP-UやCENP-Sの働きでセントロメアが動かないように制御されていると結論した。
研究の背景
生物の生命維持にとって、遺伝情報を担う染色体が正確に次世代の細胞に伝達されることは、きわめて重要である。細胞分裂期では、両極から伸びた紡錘糸が、染色体の特殊領域を捉えて、染色体を娘細胞へと分配する。この染色体の特殊領域は、セントロメアと呼ばれ、正常な染色体分配にとって必須な領域である。(前回報告「遺伝子が次世代に伝わるメカニズムの解明」の図1を参照→http://www.fbs.osaka-u.ac.jp/jpn/events/achievement/shang-fukagawa-20161104/
多くの真核生物では、セントロメアは染色体上のある特定の領域に1箇所だけ形成されるが、まれに複数の染色体領域にセントロメアが形成される場合がある。その場合には、紡錘糸が複数のセントロメア領域と結合してしまい、染色体の分断化が起きたりし、正確な染色体分配がおきなくなる。細胞は、ある特定の領域に1箇所だけセントロメアを形成する仕組みを備えていると予想される。また、一旦ある特定の領域がセントロメアとなると、次世代の細胞でも、その領域がセントロメアとして継承されると思われている。
一方で、セントロメアの形成は、特定の塩基配列上に依らずどこでも形成され得ることも分かっている。しかしながら、セントロメアは通常、反復配列上に形成されているので、実際にセントロメア領域の場所が(複数コピーある)反復配列上で固定されていて動かないかどうかは、実証することが困難であった。
深川研究室では、以前の研究においてニワトリのセントロメア領域を解析し、反復配列を含まないセントロメアを持つ染色体が存在することを見出している(Shang et al., Genome Res., 2010; Dev. Cell, 2013)。今回の研究では、反復配列を含まないセントロメアを複数の細胞で解析し、セントロメアが培養の過程で動き得るかどうかを検証した。
本研究の成果
はじめに、研究室で長く培養したニワトリDT40細胞(親株)をクローニングすることで、各1細胞から増やした独立クローン株を21種類樹立した。それぞれの独立株のセントロメアの位置について、抗セントロメア抗体を用いたChIP-seq法により高精度に解析した(図1)。
その結果、各細胞株のセントロメアの位置は、一定でなく多様な位置にセントロメアが形成されていた(図2)。
つまり、長い培養の過程でセントロメアの位置が動き、セントロメアの位置の異なる細胞の集まりが、親株のセントロメアとなっていることがわかった(図3)。
ところが、クローン化した株を再びクローン化して、それらのセントロメアの位置を調べた結果、セントロメアの位置は、比較的一定であることが判明した(図4)。
このことから、「セントロメアの位置は長い培養過程で動いてしまうが、通常は何らかのメカニズムでセントロメアが動かないように制御されている」と考えられた。
そこで、セントロメアの動きを制御している因子を探索した結果、CENP-UとCENP-Sと呼ばれるタンパク質が、関与していることを見出した。CENP-Uをノックアウトした株では、短時間の培養でもセントロメアが頻繁に動くことが観察された(図5)。
CENP-UとCENP-Sをノックアウトした細胞のセントロメア構造を解析した結果、セントロメアの構造が不安定であることが判明した。その結果を踏まえ、完全なセントロメア構造は、セントロメアの位置を固定するために重要であり、CENP-UやCENP-Sのノックアウト細胞では、不完全なセントロメア構造のため、セントロメアが動きやすくなっていると結論した(図6)。
本研究成果が社会に与える影響(本研究成果の意義)
セントロメアの数が変化したり、不安定になると、染色体が正確に分配されず、細胞に不具合が生じ、これらが、がんや多くの遺伝病の原因となることが知られている。したがって、セントロメアの形成機構を理解することは、基礎生物学的だけでなく、医学的にも重要である。
今回の研究によって、セントロメアの位置が固定される仕組みを理解できたが、その意義は大きい。セントロメアが動いてしまい、遺伝子領域にセントロメアが形成されてしまうと、その遺伝子の発現が乱れ、細胞にとって悪影響が生じる。また、セントロメアの位置の固定に関わる遺伝子の発現異常や変異が、がん細胞などでも知られており、今後はその観点からの研究も重要である。セントロメアを通じた正確な染色体分配の仕組みを理解することによって、創薬や医学応用などの研究にも発展が期待される。
特記事項
本研究は、日本学術振興会科学研究費補助金基盤研究(S)及び文科省科学研究費補助金新学術領域研究「染色体OS」の一環として行われ、国立遺伝学研究所との共同研究である。次世代シーケンス解析は、科学研究費補助金新学術領域研究「先進ゲノム」の支援を受けた。
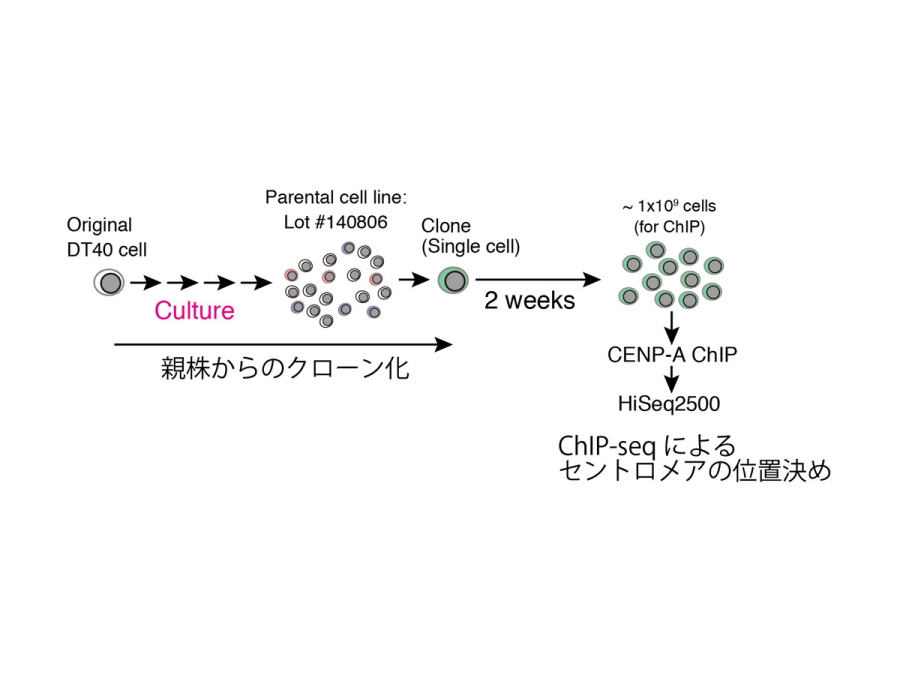
図1.実験デザイン
長期培養したDT40細胞を親株として、各1細胞に分離してクローン化した。各クローン(21種類)を2週間培養して、それぞれのセントロメアの位置をChIP-seq法により決定した。

図2.親株と21種類のクローンのセントロメアの位置
セントロメアの位置が細胞ごとに異なるのがわかる。
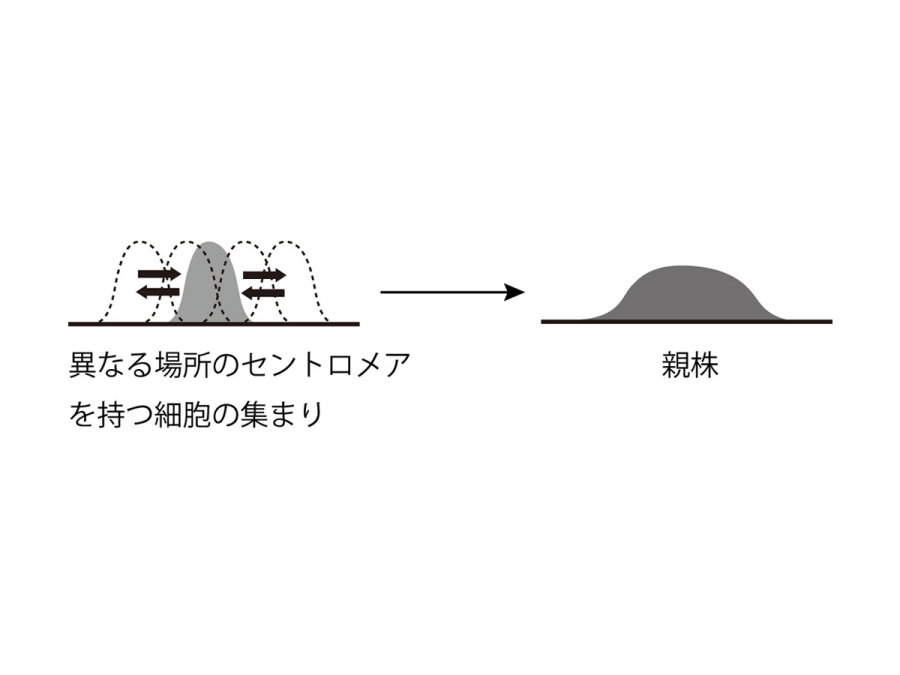
図3.親株のセントロメアが見かけ上大きく見える説明
長期培養によって、セントロメアの位置が異なる細胞が出現し、その混合が親株のセントロメアとなっている。親株のセントロメアは見かけ上大きくなっているように見える。

図4.再クローン化してすぐの各クローンのセントロメアの位置
比較的に安定に動いていない。
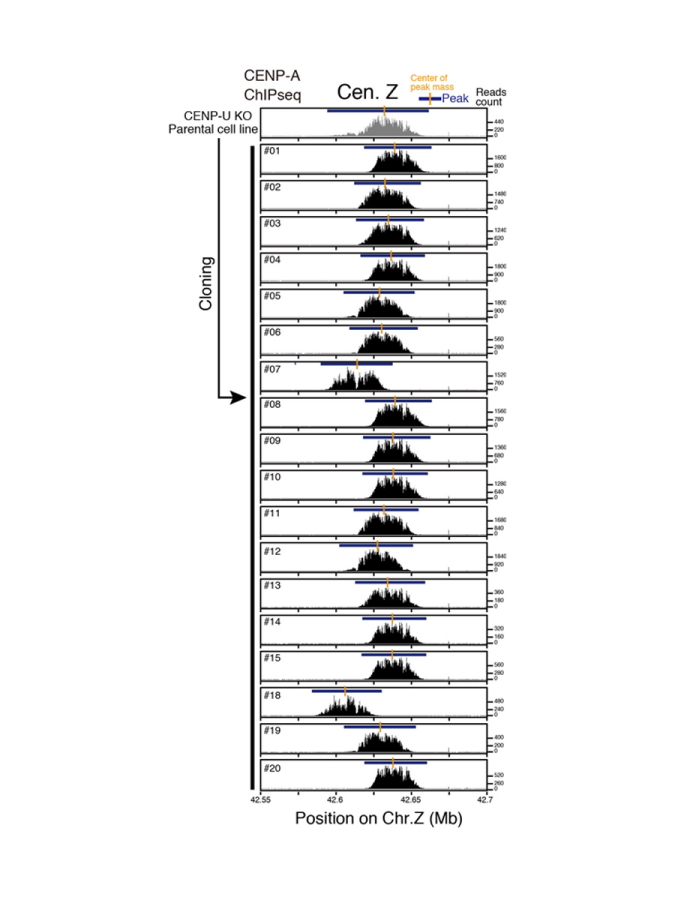
図5.CENP-Uノックアウト細胞における、クローン化直後の各セントロメアの位置
クローン化直後にもかかわらず、セントロメアがよく動いている。
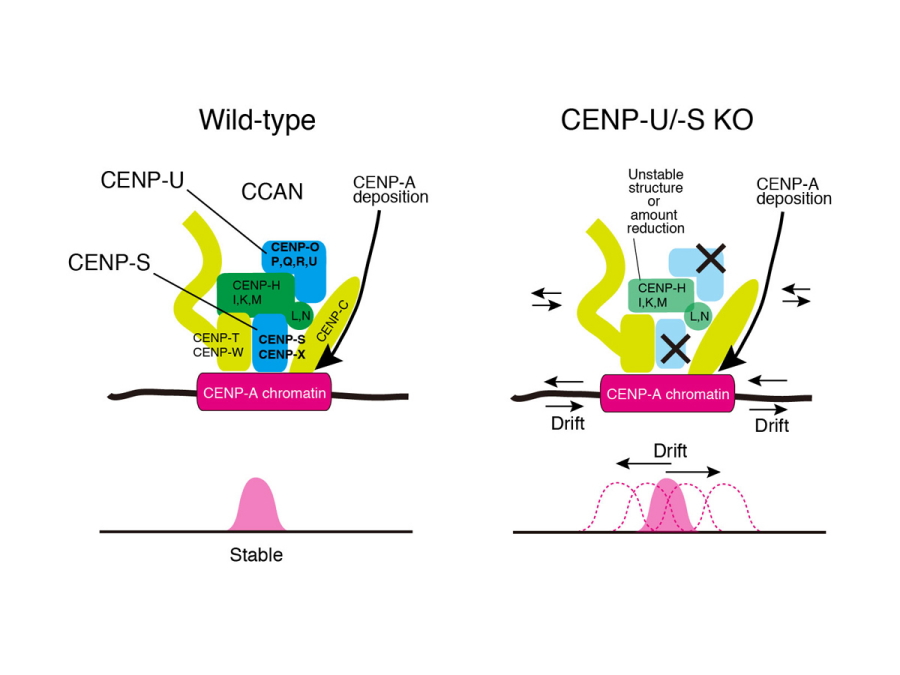
図6.本研究のまとめ
野生株では、完全なセントロメア構造が出来ており、セントロメアが動かないようにしている。一方、CENP-UやCENP-Sのノックアウト細胞では、セントロメア構造が不完全になっており、セントロメアが頻繁に動いてしまう。よって、CENP-UやCENP-Sをはじめとするセントロメアに関わるタンパク質の複合体によって、セントロメアの動きは安定化され、制御されていると考えられる。
用語解説
- セントロメア
細胞分裂の際、紡錘糸が結合する染色体の領域を指す。セントロメアの上に複数のタンパク質が集合して動原体構造を形成し、紡錘糸と結合する。どのような分子機構でセントロメア領域が規定され、その上に動原体構造が形成されるのかについては不明な点が多い。 - 反復配列
生物のゲノム上のDNA配列で、同じ配列が反復して(特に数回以上)見られるものの総称である。セントロメア領域などに見られ、ヒトのセントロメアでは、171塩基対を1ユニットとするα-サテライト呼ばれる配列が連続的に数千回繰り返されている。 - ChIP-seq法
クロマチン免疫沈降法(chromatin immunoprecipitation: ChIP)と次世代シークエンサーを組み合わせて、対象とするタンパク質の結合するDNA領域を知る技術。対象とするタンパク質に対する抗体を用いて免疫沈降した後、の回収したDNA断片を次世代シークエンサーで解析する。今回の研究では、セントロメアタンパク質CENP-Aに対する抗体で免疫沈降を行い、回収したDNAを次世代シークエンサーで解析し、ニワトリゲノムへマッピングを行った。非反復配列のセントロメアが、高精度にマップでき、セントロメアの正確な位置が決定できた。
| 原著論文 | J Cell Biol 216, 101-113 (2017) |
|---|---|
| 論文タイトル | Constitutive centromere-associated network controls centromere drift in vertebrate cells |
| 著者 | Hori T (1), Kagawa N (2), Toyoda A (3), Fujiyama A (3, 4), Misu S (5), Monma N (5), Makino F (1), Ikeo K (5), Fukagawa T (1)
|
| PubMed | 27940888 |
