細胞ネットワーク講座
染色体生物学研究室
 深川 竜郎 教授
深川 竜郎 教授
キーワード:
染色体、キネトコア、セントロメア、染色体分配
遺伝情報はどのように次世代の細胞へ伝わるのか?
我々の研究室の最も重要な関心は、「細胞分裂の過程で、染色体がどのように分配されるのか?」についてです。染色体分配の過程では、紡錘体微小管が染色体のある特殊な構造を捉えて、娘細胞に染色体を分配します。この特殊な構造は、キネトコアと呼ばれ、染色体のセントロメアという領域の上に形成されます。染色体の分配機構を理解するために、我々はキネトコア/セントロメアに注目しています。具体的には、セントロメアがゲノム領域上でどのように規定され、100種類を超えるタンパク質がキネトコアをどのように形成するのかを明らかにしようとしています。この疑問に答えるために、分子生物学、細胞生物学、生化学、構造生物学、ゲノム工学の手法を用いて研究しています。
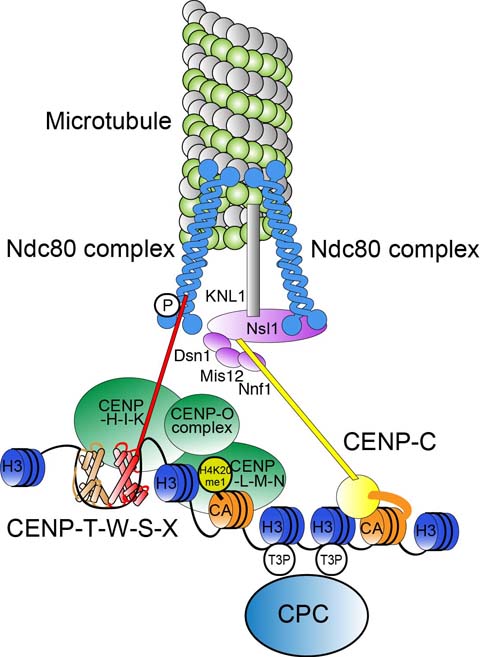
これまでに明らかになったキネトコア構造の分子模式図。この詳細の分子構造の理解と、どのようにこの構造が構築され、機能するかについて研究している。
メンバー
| 深川 竜郎 教授 | fukagawa.tatsuo.fbs[at]osaka-u.ac.jp |
|---|---|
| 堀 哲也 准教授 | t.hori.fbs[at]osaka-u.ac.jp |
| 淺川 東彦 准教授 | asakawa.haruhiko.fbs[at]osaka-u.ac.jp |
| 平野 泰弘 特任講師 | y.hirano.fbs[at]osaka-u.ac.jp |
| 有吉 眞理子 特任助教 | ariyoshi.mariko.fbs[at]osaka-u.ac.jp |
| Sridhar Shreyas 特任助教 | sridhar.shreyas.fbs[at]osaka-u.ac.jp |
| 竹之 下憂祐 特任研究員 | takenoshita.yusuke.fbs[at]osaka-u.ac.jp |
| 真鼻 裕 特任研究員 | mahana.yutaka.fbs[at]osaka-u.ac.jp |
| 牧野 文信 招へい准教授(日本電子株式会社) | |
| 久保田 佳乃 技術補佐員 | |
| 福岡 玲香 技術補佐員 |
研究者の詳細を大阪大学研究者総覧やReserach Mapで検索できます。
- ※メールアドレスの[at]は@に変換してください
Q&A
- 現在注目しているテーマは何ですか?
- 「キネトコアがどの様にできるのか?」が一番注目のテーマです。多くのキネトコア構成タンパク質が同定され、それらの再構成研究などが進んでいます。しかし、キネトコアは、細胞周期の進行とともにその構造がダイナミックに変換しますので、私たちは特にこのダイナミックな変換がどの様におこるのかを明らかにしたいと考えています。
- 最新のブレイクスルー、研究成果について教えてください。
- キネトコアの重要な働きは、微小管と染色体との橋渡しです。したがって、微小管結合タンパク質がどの様にキネトコアにリクルートされるかを明らかにすることが大切です。多くの研究者が、CENP-Cというタンパク質が、微小管結合タンパク質のリクルートに重要と考えていましたが、私たちは、CENP-Tというタンパク質の方が、CENP-Cより重要だということを明らかにしました。これは、分野の定説を覆す発見です。また、CENP-Cについては、構造解析に結果、クロマチンへの新たな結合様式を明らかにしました。
- どのようなバックグラウンドを持つメンバーで研究をすすめていますか?
- 当研究室は、これまでに、理学、農学、医学、薬学など各種の学部を卒業した人たちが所属していました。セントロメア/キネトコア研究には、多様な方法が必要なので、どの様なバックグラウンドでも活躍できます。
- 国内外の研究機関との連携について教えてください。
- 国内では、東京大学、東京工業大学、国立遺伝学研究所と密な共同研究を行っています。海外では、マサチューセッツ工科大学、エジンバラ大学、キューリ研究所などと連携して研究しています。
- 研究室から巣立った人たちはどのような道を歩まれていますか?
- 国内外の研究機関での研究職や、企業での研究職が多いです。ただし、研究以外の場で活躍している人もいます。
- 今後どんな展開が期待されますか?
- これまでは、培養細胞を用いた研究が中心でしたが、今後は個体を用いた研究や細胞ごとに異なる染色体分配機構の多様性に注目した研究を行っていこうと思っています。
研究成果
論文、総説、著書
2025年
Exploring the cell nucleus: From chromosome structure to single-cell omics
Curr. Opin. Cell Biol. 95:102530. 2025 (PMID:40381430 DOI:10.1016/j.ceb.2025.102530)
Meiosis: When centromeres choose compromise over conflict
Curr. Biol. 35(5):R196-R198. 2025 (PMID:40068619 DOI:10.1016/j.cub.2025.01.059)
Interaction mapping between nucleoporins in the fission yeast Schizosaccharomyces pombe using mass-spectrometry
J. Biochem. 177(4):273-286. 2025 (PMID:39727334 DOI:10.1093/jb/mvae095)
2024年
Molecular details and phosphoregulation of the CENP-T-Mis12 complex interaction during mitosis in DT40 cells
iScience 27(12):111295. 2024 (PMID:39628583 DOI:10.1016/j.isci.2024.111295)
CENP-C-Mis12 complex establishes a regulatory loop through Aurora B for chromosome segregation
Life Sci. Alliance 8(1):e202402927. 2024 (PMID:39433344 DOI:10.26508/lsa.202402927)
Disordered region of nuclear membrane protein Bqt4 recruits phosphatidic acid to the nuclear envelope to maintain its structural integrity
J. Biol. Chem. 300(7):107430. 2024 (PMID:38825008 DOI:10.1016/j.jbc.2024.107430)
Vertebrate centromeres in mitosis are functionally bipartite structures stabilized by cohesin
Cell 187(12):3006-3023.e26. 2024 (PMID:38744280 DOI:10.1016/j.cell.2024.04.014)
Artificial tethering of constitutive centromere-associated network proteins induces CENP-A deposition without Knl2 in DT40 cells
J. Cell Sci. 137(2): cs261639. 2024 (PMID:38319136 DOI:10.1242/jcs.261639)
2023年
An updated view of the kinetochore architecture
Trends Genet. 39(12):941-953. 2023 (PMID:37775394 DOI:10.1016/j.tig.2023.09.003)
CENP-A and CENP-B collaborate to create an open centromeric chromatin state
Nature Communu. 14(1):8227. 2023 (PMID:38086807 DOI:10.1038/s41467-023-43739-5)
Mitotic perturbation is a key mechanism of action of decitabine in myeloid tumor treatment
Cell Rep. 42(9):113098. 2023 (PMID:37714156 DOI:10.1016/j.celrep.2023.113098)
A ubiquitin-proteasome pathway degrades the inner nuclear membrane protein Bqt4 to maintain nuclear membrane homeostasis
J. Cell Sci. 136(19):jcs260930. 2023 (PMID:37694715 DOI:10.1242/jcs.260930)
Centromere/kinetochore is assembled through CENP-C oligomerization
Mol. Cell 83(13):2188-2205.e13. 2023 (PMID:37295434 DOI:10.1016/j.molcel.2023.05.023)
Ceramide synthase homolog Tlc4 maintains nuclear envelope integrity via its Golgi translocation
J. Cell Sci. 136(10):jcs260923. 2023 (PMID:37078207 DOI:10.1242/jcs.260923)
Evolutionary analysis of a complete chicken genome
Proc. Natl. Acad. Sci. USA 120(8):e2216641120. 2023 (PMID:36780517 DOI:10.1073/pnas.2216641120)
The cryo-EM structure of the CENP-A nucleosome in complex with ggKNL2
EMBO J. 42(6):e111965. 2023 (PMID:36744604 DOI:10.15252/embj.2022111965)
Transgenerational epigenetic control of constitutive heterochromatin, transposons, and centromeres
Curr. Opin. Genet. Dev. 78:102021. 2023 (PMID:36716679 DOI:10.1016/j.gde.2023.102021)
2022年
Kinetochore Architecture Employs Diverse Linker Strategies Across Evolution
Front. Cell Dev. Biol. 10:862637. 2022 (PMID:35800888 DOI:10.3389/fcell.2022.862637)
Recruitment of two Ndc80 complexes via the CENP-T pathway is sufficient for kinetochore functions
Nature Commun. 13(1):851. 2022 (PMID:35165266 DOI:10.1038/s41467-022-28403-8)
Transfected plasmid DNA is incorporated into the nucleus via nuclear envelope reformation at telophase
Commun. Biol. 5(1):78. 2022 (PMID:35058555 DOI:10.1038/s42003-022-03021-8)
Mobility of kinetochore proteins measured by FRAP analysis in living cells
Chromosome Res. 30:43-57. 2022 (PMID:34997387 DOI:10.1007/s10577-021-09678-x)
A Simple Method that Combines CRISPR and AID to Quickly Generate Conditional Knockouts for Essential Genes in Various Vertebrate Cell Lines
Methods Mol. Biol. 2377:109-122. 2022 (PMID:34709613 DOI:10.1007/978-1-0716-1720-5_6)
2021年
Bub1 and CENP-U redundantly recruit Plk1 to stabilize kinetochore-microtubule attachments and ensure accurate chromosome segregation
Cell Rep. 36(12):109740. 2021 (PMID:34551298 DOI:10.1016/j.celrep.2021.109740)
A Simple Method to Generate Super-sensitive AID (ssAID)-based Conditional Knockouts using CRISPR-based Gene Knockout in Various Vertebrate Cell Lines
Bio Protocol e11(14):e4092. 2021 (PMID:34395730 DOI:10.21769/BioProtoc.4092)
Kinetochore stretching-mediated rapid silencing of the spindle-assembly checkpoint required for failsafe chromosome segregation
Curr. Biol. 31(8):1581-1591.e3. 2021 (PMID:33651990 DOI:10.1016/j.cub.2021.01.062)
CENP-C Phosphorylation by CDK1 in vitro
Bio Protocol 11(1):e3879. 2021 (PMID:33732767 DOI:10.21769/BioProtoc.3879)
Bridgin connects the outer kinetochore to centromeric chromatin
Nature Commun. 12(1):146. 2021 (PMID:33420015 DOI:10.1038/s41467-020-20161-9)
Cryo-EM structure of the CENP-A nucleosome in complex with phosphorylated CENP-C
EMBO J. 40(5):e105671. 2021 (PMID:33463726 DOI:10.15252/embj.2020105671)
2020年
A super-sensitive auxin-inducible degron system with an engineered auxin-TIR1 pair.
Nucl. Acids. Res. 48(18):e108. 2020 (PMID:32941625 DOI:10.1093/nar/gkaa748)
H3K9me3 Maintenance on a Human Artificial Chromosome Is Required for Segregation but Not Centromere Epigenetic Memory.
J. Cell Sci. 133(14):jcs242610. 2020 (PMID:32576667 DOI:10.1242/jcs.242610)
CENP-50 Is Required for Papilloma Development in the Two-Stage Skin Carcinogenesis Model.
Cancer Sci. 111(8):2850-60 2020 (PMID:32535988 DOI:10.1111/cas.14533)
Dynamics of kinetochore structure and its regulations during mitotic progression.
Cell. Mol. Life Sci. 77(15):2981-2995. 2020 (PMID:32052088 DOI:10.1007/s00018-020-03472-4)
Artificial generation of centromeres and kinetochores to understand their structure and function.
Exp. Cell Res. 389(2):111898 2020 (PMID:32035949 DOI:10.1016/j.yexcr.2020.111898)
2019年
CDK1-mediated CENP-C phosphorylation modulates CENP-A binding and mitotic kinetochore localization
J. Cell Biol. 218(12):4042-4062 2019 (PMID:31676716 DOI:10.1083/jcb.201907006)
Cryo-EM Structures of Centromeric Tri-nucleosomes Containing a Central CENP-A Nucleosome
Structure 28(1):44-53.e4 2019 (PMID:31711756 DOI:10.1016/j.str.2019.10.016)
Centromere maintenance during DNA replication
Nat. Cell Biol. 21(6):669-671 2019 (PMID:31160706 DOI:10.1038/s41556-019-0335-0)
Where is the right path heading from the centromere to spindle microtubules?
Cell Cycle 18(11):1199-1211 2019 (PMID:31075048 DOI:10.1080/15384101.2019.1617008)
Live imaging of marked chromosome regions reveals their dynamic resolution and compaction in mitosis.
J. Cell Biol. 218(5):1531-1552 2019 (PMID:30858191 DOI:10.1083/jcb.201807125)
The CENP-A centromere targeting domain facilitates H4K20 monomethylation in the nucleosome by structural polymorphism.
Nat. Commun. 10(1):576 2019 (PMID:30718488 DOI:10.1038/s41467-019-08314-x)
2018年
Multiple phosphorylations control recruitment of the KMN-network onto kinetochores
Nat. Cell Biol. 20(12):1378-1388 2018 (PMID:30420662 DOI:10.1038/s41556-018-0230-0)
3D genomic architecture reveals that neocentromeres associate with heterochromatin regions
J. Cell Biol. 218(1):134-149 2018 (PMID:30396998 DOI:10.1083/jcb.201805003)
Cooperative Domain Formation by Homologous Motifs in HOIL-1L and SHARPIN Plays A Crucial Role in LUBAC Stabilization.
Cell Reports 23(4):1192-1204 2018 (PMID:29694895 DOI:10.1016/j.celrep.2018.03.112)
Kinetochore assembly and disassembly during mitotic entry and exit.
Curr. Opin. Cell Biol. 52:73-81 2018 (PMID:29477052 DOI:10.1016/j.ceb.2018.02.005)
Identification of PNG kinase substrates uncovers interactions with the translational repressor TRAL in the oocyte-to-embryo transition.
eLife 7:e33150 2018 (PMID:29480805 DOI:10.7554/eLife.33150)
Site-Specific Cleavage by Topoisomerase 2: A Mark of the Core Centromere.
Int. J. Mol. Sci. 19(2):E534 2018 (PMID:29439406 DOI:10.3390/ijms19020534)
2017年
Structure of the Dnmt1 Reader Module Complexed with a Unique Two-Mono-Ubiquitin Mark on Histone H3 Reveals the Basis for DNA Methylation Maintenance.
Mol. Cell 68(2):350-360.e7 2017 (PMID:29053958 DOI:10.1016/j.molcel.2017.09.037)
Critical Foundation of the Kinetochore: The Constitutive Centromere-Associated Network (CCAN).
Prog Mol Subcell Biol. 56:29-57 2017 (PMID:28840232 DOI:10.1007/978-3-319-58592-5_2)
CENP-R acts bilaterally as a tumor suppressor and as an oncogene in the two-stage skin carcinogenesis model.
Cancer Sci. 108(11):2142-2148 2017 (PMID:28795467 DOI:10.1111/cas.13348)
An efficient method to generate conditional knockout cell lines for essential genes by combination of auxin-inducible degron tag and CRISPR/Cas9.
Chromosome Res. 25(3-4):253-260 2017 (PMID:28589221 DOI:10.1007/s10577-017-9559-7)
Assembly and stoichiometry of the core structure of the bacterial flagellar type III export gate complex.
PLoS. Biol. 15(8):e2002281 2017 (PMID:28771466 DOI:10.1371/journal.pbio.2002281)
Straight and rigid flagellar hook made by insertion of the FlgG specific sequence into FlgE.
Sci Rep 7:46723 2017 (PMID:28429800 DOI:10.1038/srep46723)
Association of M18BP1/KNL2 with CENP-A nucleosome is essential for centromere formation in non-mammalian vertebrates
Dev. Cell 42(2):181-189.e3 2017 (PMID:28743004 DOI:10.1016/j.devcel.2017.06.019)
Critical histone post-translational modifications for centromere function and propagation
Cell Cycle 16(13):1259-1265 2017 (PMID:28840232 DOI:10.1080/15384101.2017.1325044)
Control of PNG kinase, a key regulator of mRNA translation, is coupled to meiosis completion at egg activation
eLife 6:e22219 2017 (PMID:28555567 DOI:10.7554/eLife.22219)
Acute inactivation of the replicative helicase in human cells triggers MCM8-9-dependent DNA synthesis
Genes Dev. 31(8):816-829 2017 (PMID:28487407 DOI:10.1101/gad.297663.117)
Stepwise unfolding supports a subunit model for vertebrate kinetochores
Proc. Natl. Acad. Sci. U. S. A. 114(12):3133-3138 2017 (PMID:28265097 DOI:10.1073/pnas.1614145114)
Constitutive centromere-associated network controls centromere drift in vertebrate cells
J. Cell Biol. 216(1):101-113 2017 (PMID:27940888 DOI:10.1083/jcb.201605001)
2016年
The architecture of the cytoplasmic region of type III secretion systems.
Sci Rep 6:33341 2016 (PMID:27686865 DOI:10.1038/srep33341)
Acetylation of histone H4 Lysine 5 and 12 is required for CENP-A deposition into centromeres
Nat. Commun. 7:13465 2016 (PMID:27811920 DOI:10.1038/ncomms13465)
Chromatin determinants of the inner-centromere rely on replication factors with functions that impart cohesion.
Oncotarget 7(42):67934-67947 2016 (PMID:27636994 DOI:10.18632/oncotarget.11982)
Kinetochore assembly and function through the cell cycle.
Chromosoma 125(4):645-59 2016 (PMID:27376724 DOI:10.1007/s00412-016-0608-3)
Chicken rRNA Gene Cluster Structure.
PLoS One 11(6):e0157464 2016 (PMID:27299357 DOI:10.1371/journal.pone.0157464)
Biochemical and Structural Analysis of Kinetochore Histone-Fold Complexes.
Methods Mol Biol. 1413:135-46 2016 (PMID:27193847 DOI:10.1007/978-1-4939-3542-0_9)
Centromeric chromatin and kinetochore assembly in vertebrate cells.
F. Hanaoka, and K. Sugasawa (eds.) DNA replication, recombination, and repair: Molecular Mechanisms and pathology 365-387 2016 ( DOI:10.1007/978-4-431-55873-6_14)
Genetic complementation analysis showed distinct contributions of the N-terminal tail of H2A.Z to epigenetic regulations.
Genes Cells 21(2):122-35 2016 (PMID:26833946 DOI:10.1111/gtc.12327)
Auxin/AID versus conventional knockouts: distinguishing the roles of CENP-T/W in mitotic kinetochore assembly and stability.
Open Biol 6(1):150230 2016 (PMID:26791246 DOI:10.1098/rsob.150230)
求める人物像(学生の方へ)
当研究室の研究内容に興味があり、研究をしたいという強い意欲のある方。生き物が好きな方、細かな手作業やモノづくりが好きな方も歓迎します。出身大学や出身学部は一切問いません。
連絡先
〒565-0871 大阪府吹田市山田丘1-3
大阪大学大学院生命機能研究科 ナノバイオロジー棟7階 染色体生物学研究室
TEL:06-6879-4428
E-mail: fukagawa.tatsuo.fbs[at]osaka-u.ac.jp(深川 竜郎 教授)
- ※メールアドレスの[at]は@に変換してください
