細菌を細胞分裂させるタンパク質が連携して働く仕組みを解明
次世代抗菌薬やマイクロマシン開発を加速させる画期的な成果
| 原著論文 | Nat. Commun. 16(1):5985 (2025) |
|---|---|
| 論文タイトル | Structural basis for the interaction between the bacterial cell division proteins FtsZ and ZapA |
| 研究室サイト | 日本電子YOKOGUSHI協働研究所〈難波 啓一 特任教授(常勤)〉 |
概要
立命館大学生命科学部の松村浩由教授、上原了助教、名古屋大学大学院理学研究科/自然科学研究機構生命創成探究センターの内橋貴之教授、大阪大学大学院生命機能研究科の難波啓一特任教授(常勤)、藤田純三特任助教(常勤)(当時)、笠井一希特任研究員の共同研究グループは、タンパク質が密集しながらもダイナミックに動き続けることで進行する、細菌の細胞分裂の巧妙な仕組みを世界で初めて解明しました。本研究では、細菌の細胞分裂において必須となるFtsZというタンパク質と、その働きを助けるZapAが連携する様子を静的な「姿(立体構造)」と動的な「動き」の両面から捉えることに成功しました。この成果は、最先端技術であるクライオ電子顕微鏡と高速原子間力顕微鏡を駆使したもので、新しい抗菌薬の精密な設計やマイクロマシン開発に道を拓くものとして期待されます。
細菌が自らを複製して増える細胞分裂は、生命の最も基本的な現象の一つです。本研究では、この細胞分裂の際に働く二種類のタンパク質が連携する巧妙な仕組みを、世界で初めて解明することに成功しました。細菌が分裂する時、まずFtsZというタンパク質が、細胞の内側で輪のような構造(Zリング)を作って細胞を内側から締め上げます。そしてZapAという別のタンパク質が、FtsZのひも(フィラメント)を束ねることを助けています。本研究は、超高性能なクライオ電子顕微鏡を用いて、FtsZとZapAが結合した状態の立体構造を原子レベルで初めて明らかにしました。さらに、高速原子間力顕微鏡によって、ZapAがFtsZに結合したり離れたりする動的なプロセスを動画として捉えることにも成功しました。タンパク質の「姿」と「動き」の両面から仕組みを解明した本成果は、細菌が増殖する根本を理解する上で非常に重要です。将来的には、この仕組みを標的として細菌の増殖を止める新しい抗菌薬の開発や、生体膜を自在に変形させる超小型機械(マイクロマシン)の創出に繋がることが期待されます。
研究の背景
地球上には、数十万種類以上もの細菌が存在し、高温や強い圧力、酸素のない場所といった、とても厳しい環境の中で、自分の体を分裂させてコピーを作り子孫を残しています。そのような過酷な環境の中で、細菌が正確に分裂するメカニズムについては不明な点が多く残されています。
本研究では、細菌の細胞分裂において主要な役割を果たすFtsZというタンパク質に着目しました。FtsZは細菌だけでなく、藻類、一部の古細菌、細胞小器官である葉緑体や一部のミトコンドリアなどに存在し、細胞や細胞小器官の分裂に関わる、生命にとって極めて重要なタンパク質です。
さらに、創薬という視点でもFtsZは重要です。細菌の一部は、黄色ブドウ球菌や肺炎桿菌(はいえんかんきん)などのように、人間に感染症を引き起こす存在です。細菌は細胞分裂して増殖することで感染を広げるため、細菌の分裂を止めることができれば、体内での細菌の数を抑えることができ、感染の進行を防ぐことが可能になります。そのため、細胞分裂において主要な役割を果たすFtsZを標的とすることで、感染症の治療に繋がると考えられます。実際に、本研究グループもこれまでFtsZを標的とした抗菌薬の種(阻害剤)を開発してきました(参考文献1、2)。
細胞の分裂は、このFtsZが細胞の真ん中に集まり、輪(リング)状の構造体を形成することから始まります(図1左)。2023年に本研究グループは、FtsZが数珠(じゅず)のように連なった「フィラメント」のような形でどのように集まるのかをクライオ電子顕微鏡によって明らかにし、「Nature Communications」に発表しました(参考文献3)。当該研究ではFtsZだけを観察対象としたため、FtsZが他のタンパク質とどのように連携して働いているのかはわかりませんでした。
FtsZのフィラメントに、本研究で着目したZapAなどの補助タンパク質が加わることで、リングはより束ねられ、細胞分裂を開始させる合図となります(参考文献4)。このリングはただ固定されているのではなく、FtsZが「GTP」とよばれるエネルギーの元になる分子を使って、一方の端で伸び、もう一方の端で分解される「トレッドミリング」という現象によって、常に動き続けているように見えます。(図1右)。
しかし、タンパク質が密集して束ねられているにもかかわらず、なぜその中でスムーズに動き続けられるのでしょうか。この謎を解き明かすため、本研究では肺炎桿菌のFtsZとZapAタンパク質を用いて、その連携の仕組みの解明に挑みました。
本研究の成果
「姿(立体構造)」の解明:クライオ電子顕微鏡による構造解析
まず「クライオ電子顕微鏡」を用いて、ZapAとFtsZが結合した複合体の様子を、原子の位置がわかるレベルの極めて高い精度(約2.7オングストローム)で立体的に観察することに成功しました(図2)。その結果、ZapAは複数のFtsZフィラメントに結合し、全体として「はしご」のような構造を形成していることが分かりました。
この構造では、2本のFtsZフィラメントと1本のFtsZフィラメントがZapAによって精巧に繋ぎ止められています。ZapAがこのようなはしご構造の横木の役割を果たすことで、FtsZフィラメントを整列させ、細胞分裂ができるように手助けしていると考えられます。また、この時、ZapAとFtsZは広い範囲で接しており、その接触がFtsZの形をわずかに変化させていることを突き止めました。
さらに、この構造から、束ねられたFtsZフィラメントの間ではマイナス電気同士の反発力が生じていることもわかりました。この反発力が、タンパク質が密集してくっついていても、自由に動くことができる一因であると考えられます。
「動き」の解明:高速原子間力顕微鏡による動態解析
次に、「高速原子間力顕微鏡」を使い、ZapAがFtsZフィラメントに結合していく様子をリアルタイムで観察しました(図3)。この観察から、ZapAが2つの異なるパターンでFtsZフィラメントを束ねていることが明らかになりました。一つは、ZapAが2本のFtsZフィラメントを跨ぐように結合するパターン、もう一つは、1本のFtsZフィラメント上で隣り合うFtsZを「繋ぐ」パターンです。これらの多様な結合パターンを使い分けることで、ZapAはFtsZフィラメントを効率よく、かつ柔軟に整列させていると考えられます。
さらに、この観察からZapAの結合が静的なものではなく、短い時間で結合と解離を活発に繰り返す、非常にダイナミックなものであることが分かりました。ZapAはFtsZフィラメントが既に2本並んでいる場所に、より長く留まる(強く結合する)傾向があることも突き止めました。また、一度ZapAが結合した場所の近くに、次のZapAが引き寄せられるように連続して結合していく「協同性」という現象も直接的に捉えることに成功しました。
研究成果のポイント
- 細菌の細胞分裂では、主要タンパク質であるFtsZと、それを助けるZapAが連携することが知られていましたが、両者がどのように結合して働くのか、その具体的な仕組みは不明でした。本研究はこの連携メカニズムを原子レベルで解明したものです。
- タンパク質の静的な立体構造を捉えるクライオ電子顕微鏡と、その動的な振る舞いを観察する高速原子間力顕微鏡。この2つの顕微鏡を組み合わせることで、FtsZとZapAが連携する仕組みを明らかにしました。
- 増殖の仕組みを原子レベルで解き明かしたことで、その働きだけをピンポイントで阻害する新しい抗菌薬の精密な設計に道を拓きます。さらに、生命の巧みな仕組みを応用し、生体膜を自在に操るマイクロマシンの開発にも繋がります。
本研究成果が社会に与える影響(本研究成果の意義)
本研究の成果は、「細胞がどのように分裂するのか」という生命の根源的な問いに、原子レベルで新たな光を当てるものです。この細胞分裂の心臓部ともいえる巧妙な仕組みを解明したことで、その働きだけをピンポイントで阻害する、副作用の少ない新しい抗菌薬の合理的な設計に道を拓きます。さらに、タンパク質が連携して膜を変形させるという生命の仕組みは、将来的に医療やバイオテクノロジーに応用可能な、超小型の機械(マイクロマシン)を創出するための重要な設計図となります。
また本成果は、静的な「姿」を捉えるクライオ電子顕微鏡と、動的な「動き」を捉える高速原子間力顕微鏡を組み合わせるアプローチが、生命の謎を解き明かす上で極めて強力であることを改めて示しました。もちろん、細菌の細胞分裂には30種類以上のタンパク質が関与しており、今回解明できたのはその複雑なシステムの一部です。本研究を重要な一歩として、今後、細胞分裂という生命現象の全貌解明に向けた研究がさらに加速することが期待されます。
特記事項
本研究成果は、2025年7月1日(現地時間)に「Nature Communications」のオンライン版で発表されました。
なお、本研究は、以下の支援のもとで実施されました。
- 松村浩由教授、藤田純三特任助教(常勤)(当時)、上原了助教、内橋貴之教授に対する科学研究費補助金(JP20K22630(J.F.)、JP23K06418(R.U.)、JP24K01994(H.M.)、JP24H02277(H.M.)、JP24H02270(H.M.)、JP23K18033(H.M.)、JP25H02292(H.M.)、JP24K0130(T.U.))
- 内橋貴之教授に対する文部科学省共同利用・共同研究システム形成事業大学等研究卓越プログラム(CURE)(補助金番号JPMXP1323015482)(T.U.)
- 松村浩由教授に対する上原記念学術振興財団 (H.M.)、長瀬科学技術振興財団 (H.M.)、ノバルティス科学振興財団 (H.M.) 、内藤科学技術振興財団 (H.M.)
- 立命館大学立命館グローバル・イノベーション研究機構R-GIRO研究プログラム(H.M.)
- 難波啓一特任教授(常勤)に対する科学技術振興機構(JST)産学共創プラットフォーム共同研究推進プログラム(OPERA:JPMJOP1861)
- 難波啓一特任教授(常勤)、松村浩由教授に対する日本医療研究開発機構(AMED)創薬等先端技術支援基盤プラットフォーム(BINDS:JP21am0101117、JP22ama121003、JP24ama121003(K.N.)、JP23ama121001(H.M.))
- 難波啓一特任教授(常勤)に対するAMED医療研究開発革新基盤創成事業(CiCLE:JP17pc0101020)、日本電子(JEOL)YOKOGUSHI協働研究所
- 上原了助教に対するG-7奨学財団、松村浩由教授に対する大阪大学蛋白質研究所拠点事業(共同研究員)(CR-20-02、CR-21-02)
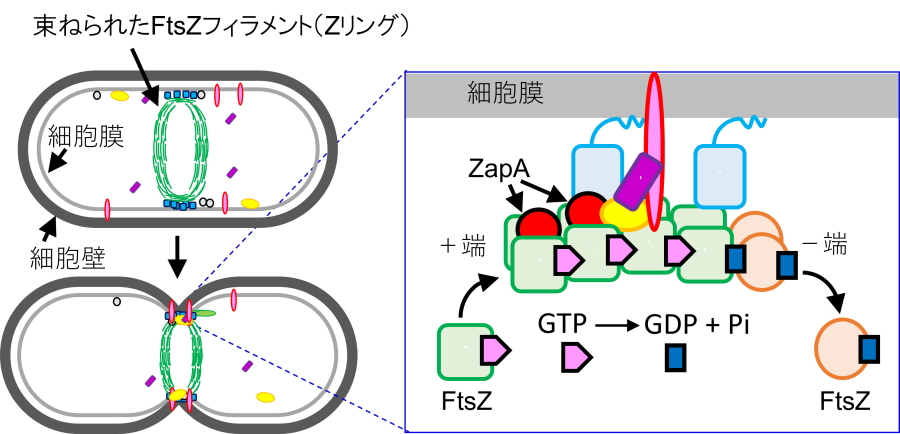
図1.細胞分裂におけるFtsZとZapAの働き
FtsZは細胞中心部でひも(フィラメント)のような形で集まり、それがZapAなどの他のタンパク質によって束ねられる。この現象が細胞分裂を開始させる合図の役割を果たしている。FtsZはGTPのエネルギーを使い、片方の端(+端)で伸びながら、もう片方の端(-端)で分解されていくというトレッドミリングを起こす(右図)。
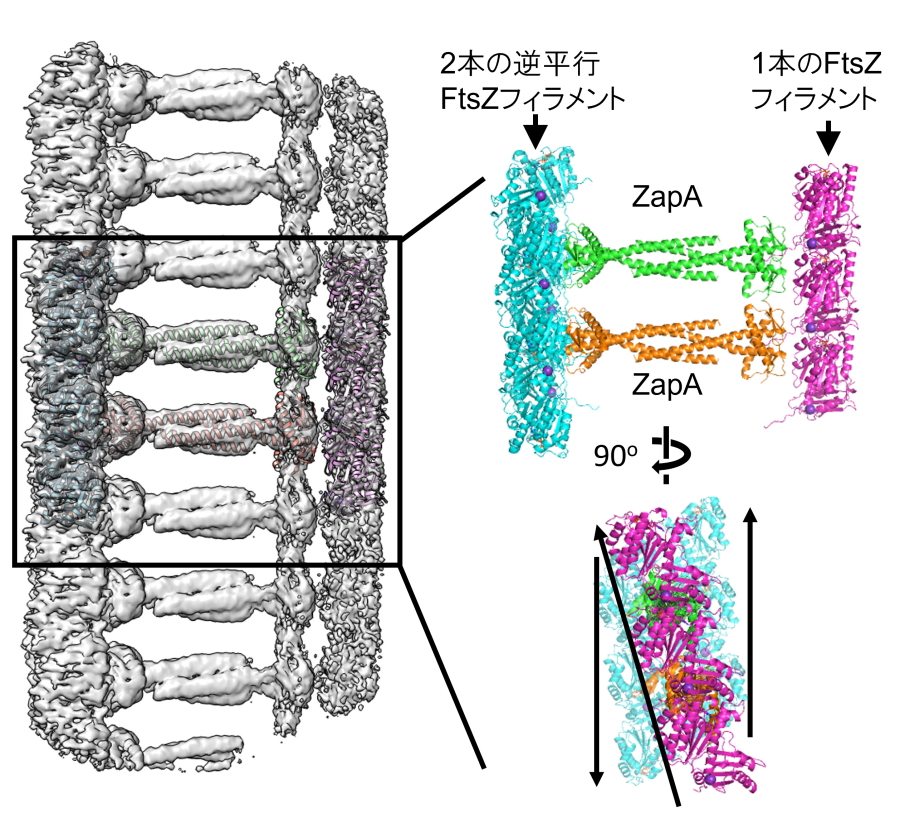
図2.クライオ電子顕微鏡で明らかとなったZapA-FtsZ複合体の立体構造
2本の逆平行FtsZフィラメントと1本のFtsZフィラメントが4量体ZapAを介して束ねられている様子が明らかとなった。
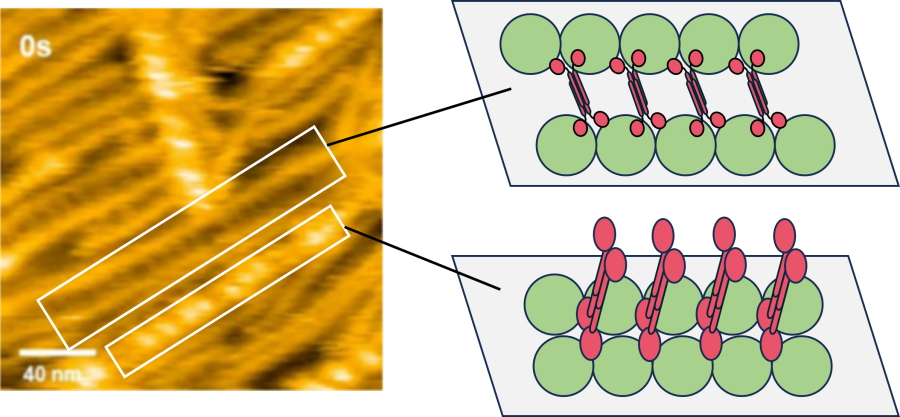
図3.ZapA-FtsZ複合体の高速原子間力顕微鏡像
ZapAが2本のFtsZフィラメントに2つの結合パターンで結合している様子が観測できた。右の模式図では、緑がFtsZ、赤がZapAを示している。2本のFtsZフィラメントを跨ぐようにしてZapAが結合する様子が確認できた。
用語解説
- クライオ電子顕微鏡
生きた状態に近い分子を急速に凍結させ、その静的な「姿(立体構造)」を原子レベルで観察できる顕微鏡技術。この技術の開発は2017年のノーベル化学賞に繋がりました。本研究では、大阪大学の難波啓一特任教授(常勤)らが開発に長年携わってきた日本電子(JEOL)社製のクライオ電子顕微鏡「CRYO ARM™ 300」が使用され、タンパク質複合体の精密な立体構造の解明に大きく貢献しました。 - 高速原子間力顕微鏡
タンパク質などが水中で活発に働く動的な「動き」を、1分子レベルでリアルタイムに捉えることができる日本発の画期的な顕微鏡技術。非常に細い針で分子の表面を優しくなぞることで、その形や動きをナノメートル(10億分の1メートル)の精度で可視化します。金沢大学の安藤敏夫特任教授らのグループによって開発され、生命科学の最前線で広く活用されています。
参考文献
- Fujita, J., et al., Structural flexibility of an inhibitor overcomes drug resistance mutations in Staphylococcus aureus FtsZ ACS Chem. Biol., 12(7), 1947-1955 (2017).
- Bryan, E., et al., Structural and antibacterial characterization of a new benzamide FtsZ inhibitor with superior bactericidal activity and in vivo efficacy against multidrug-resistant Staphylococcus aureus ACS Chem. Biol., 18, 629-642 (2023).
- Fujita, J., et al., Structures of a FtsZ single protofilament and a double-helical tube in complex with a monobody Nat. Commun., 14, 4023 (2023).
- Squyres, G.R. et al. Single-molecule imaging reveals that Z-ring condensation is essential for cell division in Bacillus subtilis. Nat. Microbiol., 6, 553-562 (2021).
| 原著論文 | Nat. Commun. 16(1):5985 (2025) |
|---|---|
| 論文タイトル | Structural basis for the interaction between the bacterial cell division proteins FtsZ and ZapA |
| 著者 | Junso Fujita (1, 2, 3), Kazuki Kasai (1, 2), Kota Hibino (4), Gota Kagoshima (5), Natsuki Kamimura (4), Shungo Tobita (4), Yuki Kato (4), Ryo Uehara (4), Keiichi Namba (1, 2), Takayuki Uchihashi (5, 6, 7), Hiroyoshi Matsumura (4)
|
| PubMed | 40593603 |
