5種類のヒト培養細胞から新たな1,074種類の難溶性RNAを同定
がんの発生や進行、ウイルスのRNAの分解などの疾患研究への基盤的なリソースとなる可能性
| 原著論文 | Nucleic Acids Res. (2023) |
|---|---|
| 論文タイトル | Landscape of semi-extractable RNAs across five human cell lines |
| 研究室サイト | RNA生体機能研究室〈廣瀬 哲郎 教授〉 |
概要
早稲田大学理工学術院総合研究所の曽超次席研究員、同大理工学術院の浜田道昭教授、熊本大学大学院生命科学研究部分子生理学講座の中條岳志講師、と大阪大学大学院生命機能研究科/理学研究科/先導的学際研究機構の廣瀬哲郎教授らの研究グループは、5種類のヒト培養細胞の独自の実験データを、新規に構築した情報解析パイプラインで解析しました。この結果、細胞内の非膜構造体形成に関与している可能性がある1,074種類の「抽出しにくいRNA(難溶性RNA)」を同定し、それらの特徴を明らかにしました。
研究の背景
細胞内制御をおこなう一つのメカニズムとして「相分離」と呼ばれる生命現象があります。これは細胞内部で、タンパク質やRNAなどが集まって液滴のような膜を持たない構造体(非膜構造体)を作る現象のことを指します。この非膜構造体は、細胞内で特定の反応を助ける「反応の場」を作るなどの役割があるとされています。
これまで、細胞内の相分離は主にタンパク質によって引き起こされると考えられていました。しかし近年の研究で、RNAもまた、凝集体の形成に重要な役割を果たすことが明らかになってきました。特にNEAT1と呼ばれるRNAは、細胞核内に「パラスペックル」と呼ばれる膜を持たない非膜構造体を作ることが知られています。
さらに近年の研究では、「抽出しにくいRNA(難溶性RNA)」という新たなRNA群が注目されています。RNA抽出法を用いて細胞を分解し遠心分離すると、水層、有機層、そして中間のタンパク層へと分けられます。これまで、細胞のRNAは全て水層に抽出されると考えられていましたが、タンパク質層に存在する抽出しにくいRNA(難溶性RNA)が存在し、細胞内の非膜構造体の形成に関与していることが知られていました。しかしながら、難溶性RNAの全体像やその特徴についてはよくわかっていませんでした。
本研究の成果
これまでの研究で同定されていた難溶性RNAは、1種類のヒト培養細胞から同定された約50種類にとどまっていました。本研究では、難溶性RNAを網羅的に同定しその特徴を解析することにより、その特徴を詳細に理解することを目的とし、5種類のヒト培養細胞から新たに1,074種類の難溶性RNAを同定しました。これらの難溶性RNAは非常に長く、リピート(反復)配列が豊富であるため、RNA結合タンパク質(RBP)を介して他の生体分子と相互作用をおこない、複雑なネットワーク構造を形成することが示唆されました。加えて、RNAの配列要素を比較する研究から、これらの難溶性RNAは4つの異なるグループに分けられ、それぞれが結合しやすいRBPの種類が異なることが分かりました(図1)。
これは、それぞれのRNAグループが異なる機能を果たしている可能性を示しています。特に、NEAT1が属するグループのRNAは、パラスペックルの形成に関わるRNA結合タンパク質と相互作用する傾向が強いことが判明しました。
本研究では第一に、難溶性RNAを抽出するための実験手法(難溶性RNA-seq)を用いて5種類のヒト培養細胞に対する実験データを取得しました。第二に、難溶性RNA-seqデータから難溶性RNAを効果的に同定するための情報解析パイプラインを構築し、得られた実験データに適用することにより難溶性RNAを網羅的に同定しました。その後、同定した難溶性RNAの細胞内の局在、クロマチンとの相互作用、構造の自由エネルギー(安定性)や長さ、GC含有率、反復配列の存在、分子間の相互作用等、多角的な特性を比較解析しました。さらに、RNA配列情報を用いた解析から、難溶性RNAを潜在的な機能の相違によって分類を行い、RBP結合モチーフ配列解析の結果、NEAT1が属するグループのRNAはパラスペックル形成要素のRBPとの結合する傾向が強いことが確認されました。
研究成果のポイント
- 細胞内で特定の反応を助ける「反応の場」となり得る「非膜構造体形成(相分離)」。近年では、タンパク質以外に、抽出しにくいRNA(難溶性RNA)がその役割を担っていると考えられているが、難溶性RNAの全体像や特徴は未解明であった。
- これまで同定されていた難溶性RNAは約50種類であったが、本研究では、5種類のヒト培養細胞の実験データを取得し、新たに1,074種類の難溶性RNAを同定した。さらに、比較解析の結果、難溶性RNAごとに結合しやすいタンパク質が異なることなどが明らかになった。これほど大規模に難溶性RNAを同定・分析した研究は世界初となる。
- タンパク質やRNAによる相分離は、疾患などのさまざまな生命現象に関与しており、今回の成果が、がんの発生や進行、ウイルスのRNAの分解、細胞のストレス反応などの研究において、基盤的なリソースとなることが期待される。
本研究成果が社会に与える影響(本研究成果の意義)
本研究は世界で初めて、多様な細胞株における難溶性RNAを同定しました。これらのRNAはそれぞれ異なる機能を有する、細胞内のさまざまな非膜構造体から派生する可能性があり、RNA中心の相分離を研究するための重要なリソースとなります。相分離は疾患などのさまざまな生命現象に関与していることが知られており、今回の研究により提供されるリソースは、がんの発生や進行、ウイルスのRNAの分解、細胞のストレス反応などの研究の基盤的なリソースとなることが期待されます。
様々なストレス状況下でストレス特異的な相分離構造体の形成が誘発されます。そのため、異なるストレス条件下でのRNAの難溶性質と潜在的な機能を調べることが今後の課題と考えています。また、難溶性RNAがどのように相分離に関係しているかの詳細メカニズムの研究も重要な課題となると思います。
研究者のコメント
近年、ノンコーディングRNAなどの機能性RNAに注目が集まっています。今回の研究結果は、細胞内構造体の形成に関与している可能性がある難溶性RNAの大規模なカタログを世界で初めて提供し、その特徴をバイオインフォマティクス的なアプローチで明らかにしました。これは、分子レベルの生命科学研究に新たな視点を提供し、将来的には疾患メカニズムの解明などにつながることを期待しています。
特記事項
本研究成果は、英国オックスフォード大学出版局によって発刊される『Nucleic Acids Research』(論文名:Landscape of semi-extractable RNAs across five human cell lines)にて、2023年7月19日10時頃(日本時間)に掲載されました。
なお、本研究は、以下の研究費の支援によって行われました。
- 科学研究費助成事業 科学研究費補助金 基盤研究(A)「リピート要素のde novo発見に基づく長鎖ノンコーディングRNAの機能の解明」(研究代表:浜田道昭(早稲田大学))
- 科学研究費助成事業 科学研究費補助金 基盤研究(A)「RNAを中心とした分子ネットワークに基づく生物学的相分離の俯瞰的・体系的理解」(研究代表:浜田道昭(早稲田大学))
- 日本医療研究開発機構(AMED)生命科学・創薬研究支援基盤事業(BINDS)「1細胞/微小組織マルチオミックスのオールインワン解析による生命科学研究の支援」(研究代表:由良敬(早稲田大学))
- 日本医療研究開発機構(AMED)次世代治療・診断実現のための創薬基盤技術開発事業(RNA標的創薬技術開発)「機能解析に基づくRNA標的創薬のための統合DBとAIシステムの構築」(研究代表:中谷和彦(大阪大学))
- 科学研究費助成事業 新学術領域研究(研究領域提案型)「『学術研究支援基盤形成』先進ゲノム解析研究推進プラットフォーム」(研究代表:黒川顕(国立遺伝学研究所))
- 日本医療研究開発機構(AMED)革新的先端研究開発支援事業「化学を基盤としたmRNAの分子設計・製造法の革新とワクチンへの展開」(研究代表:阿部洋(国立大学法人東海国立大学機構))
- 科学研究費助成事業 科学研究費基金 若手研究「Identification and characterization of lncRNAs involved in genetic compensation」(研究代表:曽超(早稲田大学))
- 科学研究費助成事業 科学研究費基金 若手研究「Identification of repetitive elements involving genome regulation」(研究代表:曽超(早稲田大学))
- 科学研究費助成事業 科学研究費補助金 学術変革領域研究(A)「非ドメイン型lncRNAの機能獲得機構の解析」(研究代表:廣瀬哲郎(大阪大学))
- 科学研究費助成事業 科学研究費補助金 挑戦的研究(萌芽)「ノンコーディングからコーディングRNAへの機能転換機構の解明」(研究代表:廣瀬哲郎(大阪大学))
- 科学技術振興機構(JST)戦略的創造研究推進事業CREST「RNAによる非膜性構造体の形成と作動原理の確立」(研究代表:廣瀬哲郎(大阪大学))
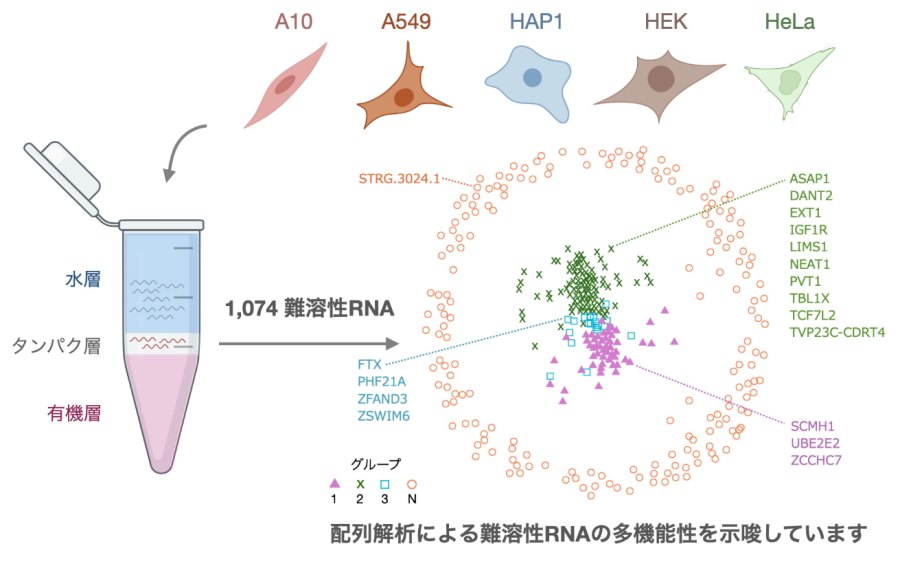
図1.配列解析による難溶性RNAの様子
本研究では、5種類のヒト培養細胞(A10、A549、HAP1、HEK、HeLa)に対する独自の実験データから、1,074種類の難溶性RNAを特定しました。さらに、難溶性RNAを塩基配列の類似性に基づいて4つのグループに分類し、それぞれが異なる機能を持つ非膜構造体から派生している可能性があることを示しました。
用語解説
- ヒト培養細胞
ヒト生体の一部を取り出し、それを特定の条件下で成長や分裂を続けさせることで、実験室内で維持される細胞のことを指します。人間の体外で細胞を生育することで、生命科学におけるさまざまな実験を可能にします。 - 情報解析パイプライン
データを集め、整理し、解析し、最終的な結果を解釈するための一連のプロセスのことを指します。これらのステップは特定の目標に応じてカスタマイズされ、組み合わされることがあります。 - 非膜構造体
脂質二重膜に覆われず、細胞内で特定の分子群を集約している構造体。混み合った細胞内空間で特定の分子を集める細胞内区画を作ることは、多数の生体分子を効率的かつ精密に制御するための生命機能の根幹をなすメカニズムである。 - パラスペックル
細胞核の中に存在する特殊な構造体の一つで、主にRNAと特定のタンパク質から成り立っています。パラスペックルはRNAの核内係留や成熟、そして転写制御に関与していると考えられています。これらの機能により、パラスペックルは細胞の正常な機能維持や病気の発症に影響を与えることが示唆されています。 - リピート(反復)配列
ゲノム中に特定の塩基配列が何度も繰り返し出現するパターンのことを指します。これらの配列は、遺伝子の機能や構造、そして種間や個体間の遺伝的多様性に影響を及ぼすことがあります。 - RNA結合タンパク質(RBP)
RBPは「RNA Binding Protein」の略で、RNAと結合する性質を持つタンパク質のことを指します。これらのタンパク質は、RNAの構造形成、遺伝子の発現調節、細胞内でのRNAの位置決定など、RNAが関与する生物学的プロセスにおいて重要な役割を果たします。 - 難溶性RNA-seq
一般的な溶媒やバッファーでは取り出しにくい特殊なRNA、つまり難溶性RNAに焦点を当てたRNAシーケンシングの一種です。RNAシーケンシングとは、RNAの塩基配列を大規模に解析する技術のことを指します。 - クロマチン
DNAとそれに結合した特殊なタンパク質(ヒストンなど)から構成される複雑な構造を指します。細胞の核内に存在し、DNAをコンパクトに折りたたんで収納する役割があります。クロマチンの形状や構造が変わることで、遺伝情報の読み取りやコピー、修復が調節されます。 - 構造の自由エネルギー
RNA分子が特定の構造を持つために必要なエネルギーを意味します。このエネルギーはRNAの折りたたみ、つまりRNAが特定の形状になるプロセスを支配します。RNAの構造は、基本的に塩基配列によって決まります。それぞれの塩基配列が形成する特定の構造は、その安定性(エネルギー状態)によって決まります。エネルギーが低いほど、その構造はより安定となります。 - GC含有率
RNA分子全体の中でグアニン(G)とシトシン(C)の合計が占める割合のことを指します。RNAは4種類のヌクレオチド(アデニン[A]、グアニン[G]、シトシン[C]、ウラシル[U])から構成されており、GC含有率はこれらのうちGとCの割合を示しています。この値は、RNAの物理的、化学的性質や生物学的機能に影響を及ぼすことがあります。 - RBP結合モチーフ
RBP(RNA結合タンパク質)が特定のRNA配列または構造に結合するための特異的な配列または構造パターンのことを指します。RBP結合モチーフは、RNAとタンパク質との相互作用の基盤を形成し、細胞内のRNAの動きと役割を調整します。 - ノンコーディングRNA
タンパク質をコードしないRNAのことを指します。一般的に、DNAが遺伝情報を持ち、RNAがその情報を使ってタンパク質を生産すると考えられています。しかし、すべてのRNAがタンパク質を作るわけではありません。タンパク質を作らないRNAがノンコーディングRNAです。これらは遺伝情報の調節や遺伝子の発現に関与し、細胞の生命活動に重要な役割を果たします。
| 原著論文 | Nucleic Acids Res. (2023) |
|---|---|
| 論文タイトル | Landscape of semi-extractable RNAs across five human cell lines |
| 著者 | Chao Zeng (1), Takeshi Chujo (2), Tetsuro Hirose (3, 4), Michiaki Hamada (1, 5, 6)
|
