細胞ネットワーク講座
RNA生体機能研究室
 廣瀬 哲郎 教授
廣瀬 哲郎 教授
キーワード:
ノンコーディングRNA、相分離、非膜性構造体、ストレス応答、疾患
細胞内構造体を構築するノンコーディングRNAの作用機構について研究しています
今世紀初頭のポストゲノム解析によって、真核生物ゲノムの大部分を占める非コード領域から大量のノンコーディングRNA(ncRNA)が産生されていることが明らかになり、その機能に大きな注目が集まっています。私たちの研究室では、ncRNAの生体機能を明らかにし、その働きを規定する新たな遺伝暗号ルールを解明することによって、ゲノム機能概念を再構築することを目指しています。特に、これまで私たちが明らかにしてきたncRNAが誘導する相分離現象による細胞内構造体の形成機構やその役割について、基盤的な分子・細胞生物学研究に生物物理学や情報科学などの手法を取り込んで研究しています。
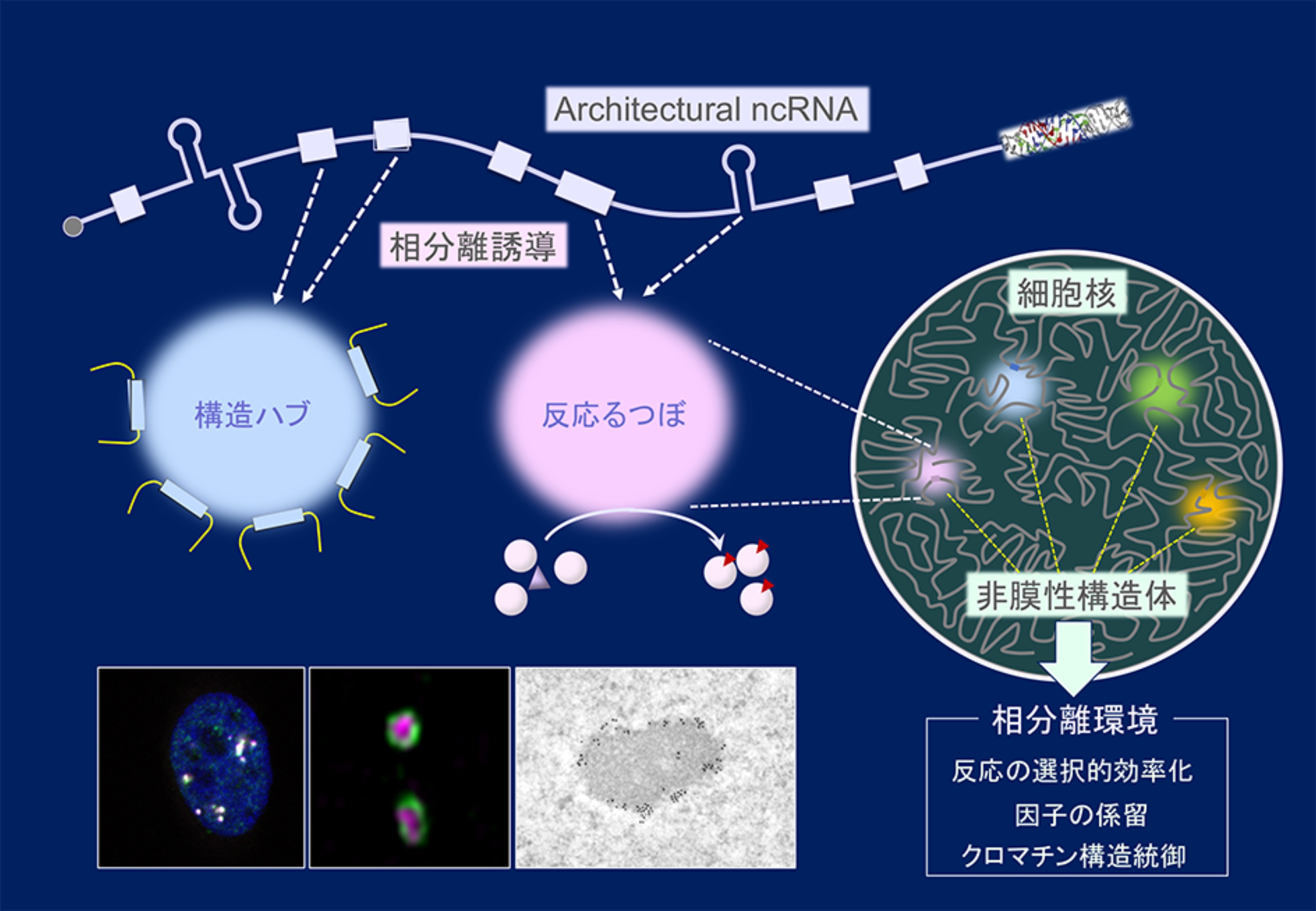
Architectural ncRNAは相分離を介して非膜性構造体を形成する。このncRNAは独自の機能ドメインを持ち、細胞内の構造ハブや生化学反応の「るつぼ」として機能している。顕微鏡写真は左からncRNA依存的非膜性構造体パラスペックルの共焦点顕微鏡(左)、超解像顕微鏡(中央)、電子顕微鏡(右)によって得られたものである。
メンバー
| 廣瀬 哲郎 教授 | hirose.tetsuro.fbs[at]osaka-u.ac.jp |
|---|---|
| 山崎 智弘 准教授 | tyamazaki[at]fbs.osaka-u.ac.jp |
| 二宮 賢介 特任講師(常勤) | ninomiya.kensuke.fbs[at]osaka-u.ac.jp |
| 谷口 一郎 特任助教(常勤) | taniguchi.ichiro.fbs[at]osaka-u.ac.jp |
| 北 加奈子 助教(医学系研究科) | |
| 藤原 奈央子 助教 | nfujiwara.fbs[at]osaka-u.ac.jp |
| 高橋 利恵子 技術補佐員 | |
| 淺田久子 特任技術職員 | |
| 江副 香奈子 事務補佐員(秘書) | |
研究者の詳細を大阪大学研究者総覧やResearch Mapで検索できます。
- ※メールアドレスの[at]は@に変換してください
Q&A
- 現在注目しているテーマは何ですか?
- ncRNA暗号の解読
- ncRNAによる相分離や相転移の誘導機構
- ncRNA構造体とクロマチンの相互作用機構
- ゲノム中のリピート配列の機能
- 種特異性の獲得機構
- 最新のブレイクスルー、研究成果について教えてください。
- 数万種類にも及ぶncRNA群の中から細胞内の非膜性構造体の骨格として働くncRNAを発見しました。その後、そのncRNAが多くの天然変性タンパク質を集約して細胞内相分離を誘発し、これによって構造体を形成していること、またヒトゲノムから類似機能を持つ数多くのncRNAが産生されていることを明らかにしました。こうした結果をもとに、これらのncRNA群を新たなncRNA機能カテゴリーとして「architectural ncRNA」と呼ぶことを提唱しました。
- どのようなバックグラウンドを持つメンバーで研究をすすめていますか?
- これまでの研究室メンバーの出身学部は、理学部、工学部、農学部、医学部など多岐にわたっています。
- 国内外の研究機関との連携について教えてください。
- 国内では北大、東大、産総研、理研など、海外ではCNRS(仏)、マックスプランク研究所(独)、フランシスクリック研究所(英)、ロチェスター大学(米)、西オーストラリア大学(豪)、KAUST(サウジアラビア)など
- 研究室から巣立った人たちはどのような道を歩まれていますか?
- 多くの人がアカデミアポジション(熊本大学助教、立命館大学助教、国際医療研究センター上級研究員、アリゾナ大学ポスドクなど)やバイオベンチャー企業などに進みました。
- 今後どんな展開が期待されますか?
- ncRNA機能理解とそれを担う新暗号の解読によって、「ゲノムの働き方」の概念を再構築できると期待されます。さらにncRNA機能が関わる様々な生理現象や疾患発症機構に関する新しい視点での理解が進むと考えられます。
- 最近の代表論文
- Hirose et al. (2022) Nat Rev Mol Cell Biol.
Ninomiya et al. (2021) EMBO J. 40, e107976
Yamazaki et al. (2021) EMBO J. 40, e107270
Ninomiya et al. (2020) EMBO J. 39, e102729
Yamazaki et al. (2018) Mol. Cell 70, 1038-1053
Chujo et al. (2017) EMBO J. 30, 1447-1462
Mannen et al. (2016) J. Cell Biol. 214, 45-59
Kawaguchi et al. (2015) PNAS 112, 4304-4309
研究成果
論文、総説、著書
2023年
Functional domains of nuclear long noncoding RNAs: Insights into gene regulation and intracellular architecture
Curr Opin Cell Biol. 5:102250 2023 (PMID:37806294 DOI:10.1016/j.ceb.2023.102250.)
Nascent ribosomal RNA act as surfactant that suppresses growth of fibrillar centers in nucleolus.
Commun Biol 6(1):1129 2023 (PMID:37935838 DOI:10.1038/s42003-023-05519-1)
Shell protein composition specified by the lncRNA NEAT1 domains dictates the formation of paraspeckles as distinct membraneless organelles
Nat. Cell Biol. 25(11):1664-1675 2023 (PMID:37932453 DOI:10.1038/s41556-023-01254-1)
Satellite RNAs: emerging players in subnuclear architecture and gene regulation
EMBO J. 42(18):e114331 2023 (PMID:37526230 DOI:10.15252/embj.2023114331)
Nondomain biopolymers: Flexible molecular strategies to acquire biological functions
Genes Cells. 28(8):539-552 2023 (PMID:37249032 DOI:10.1111/gtc.13050)
Landscape of semi-extractable RNAs across five human cell lines
Nucleic Acids Res. 51(15):7820-7831 2023 (PMID:37463833 DOI:10.1093/nar/gkad567)
A guide to membraneless organelles and their various roles in gene regulation
Nat Rev Mol Cell Biol. 24(4):288-304 2023 (PMID:36424481 DOI:10.1038/s41580-022-00558-8)
Remarkable improvement in detection of readthrough downstream-of-gene transcripts by semi-extractable RNA-sequencing
RNA. 29(2):170-177 2023 (PMID:36384963 DOI:10.1261/rna.079469.122)
Long non-coding RNAs: definitions, functions, challenges and recommendations
Nat Rev Mol Cell Biol. 24(6):430-447 2023 (PMID:36596869 DOI:10.1038/s41580-022-00566-8)
2022年
tRNA-like Transcripts from the NEAT1-MALAT1 Genomic Region Critically Influence Human Innate Immunity and Macrophage Functions
Cells. 11(24):3970 2022 (PMID:36552736 DOI:10.3390/cells11243970)
Triblock copolymer micelle model of spherical paraspeckles
Front Mol Biosci. 9:925058 2022 (PMID:36072433 DOI:10.3389/fmolb.2022.925058)
Micellization: A new principle in the formation of biomolecular condensates
Front Mol Biosci. 9:974772 2022 (PMID:36106018 DOI:10.3389/fmolb.2022.974772)
Statistical Thermodynamics Approach for Intracellular Phase Separation
Methods Mol Biol. 2509:361-393 2022 (PMID:35796975 DOI:10.1007/978-1-0716-2380-0_22)
lncRNA Neat1 regulates neuronal dysfunction post-sepsis via stabilization of hemoglobin subunit beta
Mol Ther. 30(7):2618-2632 2022 (PMID:35331906 DOI:10.1016/j.ymthe.2022.03.011)
2021年
NEAT1 is essential for metabolic changes that promote breast cancer growth and metastasis
Cell Metab. 33(12):2380-2397 2021 (PMID:34879239 DOI:10.1016/j.cmet.2021.11.011)
Noncoding RNAs: biology and applications-a Keystone Symposia report
Ann N Y Acad Sci 1506(1):118-141 2021 (PMID:34791665 DOI:10.1111/nyas.14713)
Distinct RNA polymerase transcripts direct the assembly of phase-separated DBC1 nuclear bodies in different cell lines
Mol Biol Cell. 32(21):ar33 2021 (PMID:34495685 DOI:10.1091/mbc.e21-02-0081)
SPF45/RBM17-dependent, but not U2AF-dependent, splicing in a distinct subset of human short introns
Nat Commun. 12(1):4910 2021 (PMID:34389706 DOI:10.1038/s41467-021-24879-y)
Control of condensates dictates nucleolar architecture
Science. 373(6554):486-487 2021 (PMID:34326220 DOI:10.1126/science.abj8350)
m6A modification of HSATIII lncRNAs regulates temperature-dependent splicing
EMBO J. 40(15) e107976 2021 (PMID:34184765 DOI:10.15252/embj.2021107976)
ArcRNAs and the formation of nuclear bodies
Mamm genome. 33(2):382-401 2021 (PMID:34085114 DOI:10.1007/s00335-021-09881-5)
Paraspeckles are constructed as block copolymer micelles
EMBO J. 40(12) e107270 2021 (PMID:33885174 DOI:10.15252/embj.2020107270)
Small molecule targeting r(UGGAA)n disrupts RNA foci and alleviates disease phenotype in Drosophila model
Nat Commun. 12(1):236-236 2021 (PMID:33431896 DOI:10.1038/s41467-020-20487-4)
CRISPR-Mediated Mutagenesis of Long Noncoding RNAs
Methods Mol Biol. 2254:283-303 2021 (PMID:33326083 DOI:10.1007/978-1-0716-1158-6_18)
2020年
Inhibition of the long non-coding RNA NEAT1 protects cardiomyocytes from hypoxia in vitro via decreased pri-miRNA processing
Cell Death Dis. 11(8):677 2020 (PMID:32826883 DOI:10.1038/s41419-020-02854-7)
Functional annotation of human long noncoding RNAs via molecular phenotyping
Genome Res. 30(7):1060-1072 2020 (PMID:32718982 DOI:10.1101/gr.254219.119)
NONO Is a Negative Regulator of SOX2 Promoter
Cancer Genomics Proteomics. 17(4):359-367 2020 (PMID:32576581 DOI:10.21873/cgp.20195)
Long non-coding RNA Neat1 regulates adaptive behavioural response to stress in mice
Transl Psychiatry 10(1):171-171 2020 (PMID:32467583 DOI:10.1038/s41398-020-0854-2)
Phase separation driven by production of architectural RNA transcripts
Soft Matter. 16(19):4692-4698 2020 (PMID:32396591 DOI:10.1039/c9sm02458a)
Detailed analyses of the crucial functions of Zn transporter proteins in alkaline phosphatase activation
Journal of Biological Chamistry 295(17):5669-5684 2020 (PMID:32179649 DOI:10.1074/jbc.RA120.012610)
求める人物像(学生の方へ)
当研究室の研究内容に興味があり、研究をしたいという強い意欲のある方。生き物が好きな方、細かな手作業やモノづくりが好きな方も歓迎します。出身大学や出身学部は一切問いません。
連絡先
〒565-0871 大阪府吹田市山田丘1-3
大阪大学大学院生命機能研究科 生命機能A棟1階 RNA生体機能研究室
TEL: 06-6879-4675
E-mail: hirose.tetsuro.fbs[at]osaka-u.ac.jp(廣瀬 哲郎 教授)
- ※メールアドレスの[at]は@に変換してください
