凝集したタンパク質を元に戻す分子Hsp104の構造を解明
| 原著論文 | Structure 29:1–10 (2021) |
|---|---|
| 論文タイトル | Split conformation of Chaetomium thermophilum Hsp104 disaggregase |
| 研究室サイト | 日本電子YOKOGUSHI協働研究所〈難波 啓一 特任教授(常勤)〉 |
概要
大阪大学大学院生命機能研究科の川本晃大特任助教(常勤)(現・大阪大学蛋白質研究所 助教)、難波啓一特任教授(理化学研究所生命機能科学研究センター超分子システム動態研究チーム チームリーダー、放射光科学研究センター 副センター長・兼任)、東京農工大学工学府生命工学専攻の井上耀介さん(博士前期課程・当時)、東京農工大学工学研究院生命機能科学部門の篠原恭介准教授、養王田正文教授、野口恵一教授、京都大学大学院理学研究科の花園祐矢研究員(現・国立研究開発法人量子科学技術研究開発機構)、竹田一旗准教授、三木邦夫教授、熊本大学発生医学研究所の野井健太郎研究員(現・大阪大学ナノサイエンスデザイン教育研究センター 特任助教(常勤))、小椋光教授(現・熊本大学大学院生命科学研究部)、筑波大学計算科学研究センターの重田育照教授らのグループは、細胞内で凝集したタンパク質を再生する分子Hsp104の構造を明らかにしました。X線結晶構造解析・クライオ電子顕微鏡解析・高速原子間力顕微鏡(高速AFM)の技術を用いてこれまでに知られていなかったHsp104の構造を決定しました。この成果により今後、タンパク質の異常凝集が引き起こすと考えられている神経変性疾患など病気の基礎的な理解へ向けた貢献が期待されます。
研究の背景
Hsp104は真核生物の細胞内で変性凝集してしまったタンパク質を元の立体構造へ戻すことのできる分子シャペロンです。近年、組織内に異常凝集したタンパク質線維を生じるパーキンソン病のモデル動物に対してHsp104を注入すると症状が改善するという報告があり神経変性疾患の治療において注目されています。これまでの研究からHsp104は6量体のリングを形成することが分かっていました。また凝集したタンパク質を捕まえ元に戻す仕組みとして以下のことが考えられています。初めにリング内部で凝集したタンパク質の末端と結合しアデノシン三リン酸(ATP)の加水分解反応に伴う構造変化が起こります。次にその構造変化を駆動力として捕捉した基質末端を連続的に手繰り寄せることで凝集したタンパク質をポリペプチド鎖に変換するというモデルです。これはクライオ電子顕微鏡を用いた構造解析の知見を元に提案されています。一方で、Hsp104が基質である凝集タンパク質と反応する前の状態は詳しく分かっていませんでした。
本研究の成果
Hsp104の詳しい構造を明らかにするため、安定性が高く構造解析に適したタンパク質を生産することで知られる好熱性真菌由来のHsp104を大腸菌の組み換え体として獲得しました。構造解析のために準備した数種類のタンパク質の中で検討した結果、アデノシン二リン酸(ADP)存在下のN末端を欠損する変異体(Hsp104ΔN)においてX線結晶構造解析により詳しい構造を決定することに成功しました。ここで得られた構造はこれまで報告されてきたリング状の形態ではなく、6つのサブユニットで構成される基本単位の中の1番目と6番目のサブユニットが物理的に接触しない開いたらせん構造をとっていました。次に、この「開いたらせん構造」の妥当性を検証するため、もうひとつの高分解能構造を決める手法であるクライオ電子顕微鏡解析により同じくN末端欠損変異体の構造決定を試みました。その結果、1番目と6番目のサブユニットが物理的に接触している「閉じたリング構造」と接触していない「開いたらせん構造」、それぞれを有する2種類の6量体構造を決定することに成功しました。前者の「閉じたリング構造」を有するHsp104の6量体は先行研究で海外のグループが報告していましたが、後者の「開いたらせん構造」を有する6量体はこれまで知られていなかった新しい構造であると言えます。Hsp104の反応サイクルを考えるとADPと結合したHsp104の構造は加水分解反応後の基質(凝集したタンパク質)と反応していない状態の構造であると予想されます。このことを検証するために、タンパク質の構造の変化をリアルタイムで捉えることが可能な高速原子間力顕微鏡(高速AFM)を用いてこの「開いたらせん構造」がHsp104の反応の中でいつどのようにして現れるかを検証しました。全長を有する野生型Hsp104を用いて観察を行いました。ADPの存在下でHsp104を経時的に観察すると、「閉じたリング構造」と「開いたらせん構造」の2種類の構造が繰返し観察されました。この溶液条件では加水分解反応を伴わないことから、「閉じたリング構造」と「開いたらせん構造」間の変化は、加水分解反応と共役した能動的なものではなくHsp104が内在的に有する柔軟性に起因する確率的に起こる構造変化であることが示唆されました。最後に、実験系にHsp104のモデル基質であるカゼインを添加し加水分解反応が起こるATP存在下でHsp104がどのような振る舞いを示すか調べました。その結果、「中央の孔が無い構造」と「開いたらせん構造」のそれぞれ異なる構造が観察されました。前者は基質をリング中央に補捉し反応している最中の状態、後者は基質と反応していない状態の構造であると考えられます。基質を添加しない条件と比べて基質を添加した条件では「開いたらせん構造」の出現頻度が増加しました。これらのことから、今回見つかった「開いたらせん構造」はHsp104が基質を補捉し反応する状態以外の、基質と出会う前の状態であると考えられます。
本研究成果が社会に与える影響(本研究成果の意義)
今回得られた成果は、まだ詳細が分かっていないHsp104が基質(凝集したタンパク質)を認識し捕捉するしくみを明らかにする手がかりとなるものであると考えています。今後、さらに研究をすすめることで基質の一つである病気の原因となるタンパク質凝集線維とHsp104が反応するしくみの解明や反応効率の高い改変型Hsp104の開発につながることが期待されます。
特記事項
本研究成果は、2021年3月1日(月)に米国科学誌「Structure」(オンライン)に掲載されました。
本研究は、大阪大学の難波啓一特任教授(大学院生命機能研究科)のグループと東京農工大学の篠原恭介准教授(大学院工学研究院/グローバルイノベーション研究院)、京都大学の三木邦夫教授(大学院理学研究科)、熊本大学の小椋光教授(発生医学研究所)、筑波大学の重田育照教授(計算科学研究センター)との共同研究により実施されました。
本研究は科学研究費補助金新学術領域「分子夾雑の生命化学」(公募研究18H04541・篠原恭介)、熊本大学発生医学研究所共同研究支援(篠原恭介・養王田正文)、科学技術振興機構・戦略的創造研究推進事業CREST(JPMJCR13M1・小椋光)と日本医療研究開発機構・創薬等先端技術支援基盤プラットフォームAMED BINDS(JP19am0101117・難波啓一)の支援を受け実施されました。
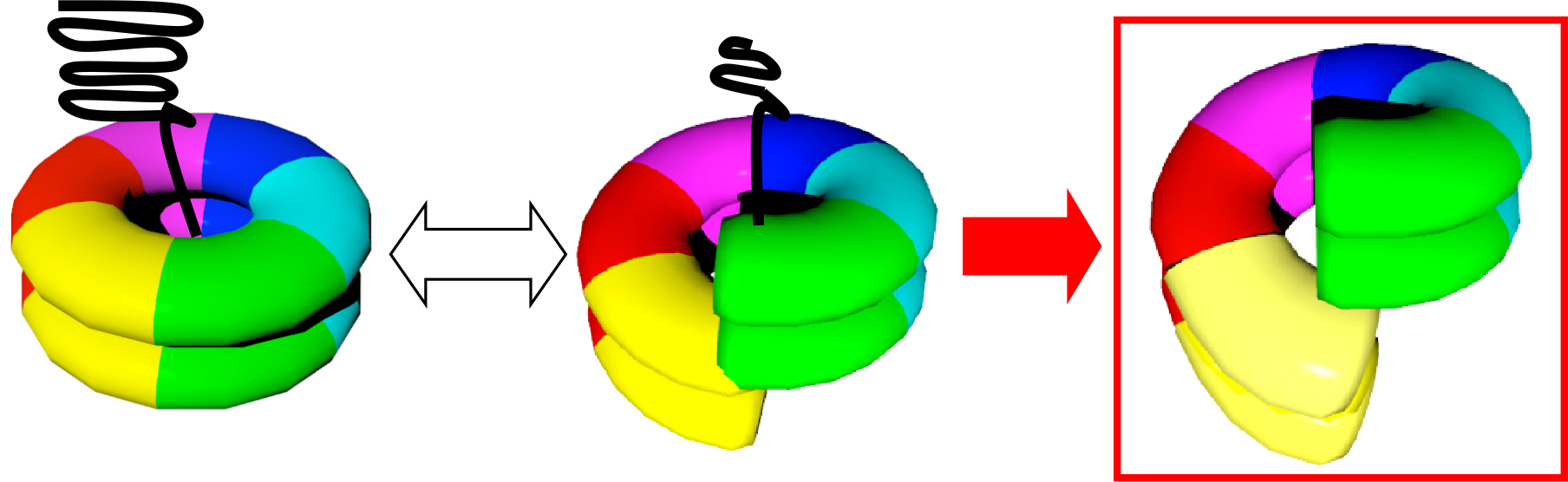
図1.Hsp104の反応サイクルと構造変化
今回見つけた基質と反応していない状態と想定される構造(赤の四角)
| 原著論文 | Structure 29:1–10 (2021) |
|---|---|
| 論文タイトル | Split conformation of Chaetomium thermophilum Hsp104 disaggregase |
| 著者 | Yosuke Inoue (1), Yuya Hanazono (2), Kentaro Noi (3), Akihiro Kawamoto (4), Masato Kimatsuka (5), Ryuhei Harada (5), Kazuki Takeda (2), Ryoichi Kita (1), Natsuki Iwamasa (1), Kyoka Shibata (1), Keiichi Noguchi (1), Yasuteru Shigeta (5), Keiichi Namba (4, 6, 7), Teru Ogura (3), Kunio Miki (2), Kyosuke Shinohara (1), Masafumi Yohda (1)
|
| PubMed | 33651974 |
