神経結合は胎生期の遺伝子発現制御によりあらかじめ規定される。
| 原著論文 | BMC Biol 14, 103 (2016) |
|---|---|
| 論文タイトル | Establishment of high reciprocal connectivity between clonal cortical neurons is regulated by the Dnmt3b DNA methyltransferase and clustered protocadherins |
| 研究室サイト | 心生物学研究室〈八木 健 教授〉 |
概要
大脳皮質が正常な機能を発揮するには、神経細胞同士が適切な相手とシナプス結合することが重要です。これまでに、胎生期に同じ神経幹細胞から生まれた神経細胞群は生後シナプス結合を作りやすいことが報告されており、この結合は発達初期の脳機能に重要と考えられています。今回、生理学研究所の足澤悦子特任助教と吉村由美子教授は、同じく生理学研究所の平林真澄准教授と大阪大学大学院生命機能研究科の八木健教授との共同研究によって、マウスを用いて同じ神経幹細胞から生まれた神経細胞がシナプス結合する過程を詳しく調べた結果、同じ神経幹細胞から生まれた神経細胞同士が優先的にシナプス結合を形成しあっていることを証明しました。胎生期にのみ発現する、遺伝子の発現を制御する酵素である「DNAメチル化酵素」の働きが、将来的にシナプス結合を作るか否かの手がかりを作り、その制御を受ける細胞接着因子「クラスター型プロトカドヘリン」が、シナプス結合する相手を認識するのに重要であることを明らかにしました。
本研究結果は、英国科学雑誌であるBMC Biology誌に掲載されました(日本時間12月2日オンライン版掲載)。
研究の背景
脳の正常な発達は、遺伝的にネットワークを形成するようにプログラミングされた神経細胞同士の結合(シナプス結合)から始まります。これまでの先行研究では、胎生期に同じ神経幹細胞から生まれた姉妹神経細胞同士(図1)は、生後の発達過程においてシナプス結合を形成しやすいことが報告されていました。つまり、生後のシナプス結合の形成には、胎生期の遺伝子プログラミングが深く関与していると考えられるのですが、その詳しい遺伝子制御や、分子メカニズムについては、これまでよく分かっていませんでした。
本研究の成果
今回私たちは、生後発達期のマウスの大脳皮質体性感覚野に着目し、この領域のシナプス結合を詳細に解析しました(図1)。結果、二つの姉妹細胞がお互いにシナプス結合を形成する双方向性シナプス結合の関係にあることを発見しました(図2)。これまでの先行研究によって、双方向性シナプス結合している神経細胞ペアは同じ情報を共有していることが明らかにされていることからも、この双方向性シナプス結合は脳内の情報処理メカニズムにおいて大変重要な結合と考えられています。
さらに胎生期にのみ存在し、遺伝子の発現を制御する酵素である「DNAメチル化酵素(Dnmt3b)」による遺伝子発現制御が、双方向性シナプス結合の形成に必要であることがこのDnmt3bの遺伝子を欠損させたマウスのシナプス結合を解析することにより明らかになりました(図2)。つまりこの酵素は、胎生期の大脳皮質の発生初期にしか働いていないにも関わらず、胎生期の遺伝子発現制御が生後のシナプス結合形成にまで影響を与えているということになります。これは生後の神経ネットワークの形成に、胎生期の遺伝子発現制御が関わることを世界で初めて証明した、驚くべき発見と言えます。
さらに、このDnmt3bは数百種類の遺伝子の発現を制御することが知られていますが、今回の私たちの研究では、Dnmt3bが制御する膨大な量の遺伝子発現の中から、特に姉妹細胞間の双方向性シナプス結合の形成に関わる重要な分子の同定に成功しました。共同研究者である八木教授らの研究グループが発見したクラスター型プロトカドヘリンは58種類の分子からなり、私たちの体をつくる分子の中でもっとも種類が多い細胞接着分子です。個々の神経細胞において、この58種類の中から選ばれた約15種類が発現しており、その15種類の選択にDnmt3bが関わることも八木教授らの研究グループにより明らかにされています。この組み合わせは個々の神経細胞で異なることから、組み合わせのパターンによって神経細胞に個性を持たせていると考えられています。しかし、その多様な発現パターンがどのような働きをしているかについては、これまでほとんど明らかにされていませんでした。
今回私たちは、このクラスター型プロトカドヘリンを欠損させた姉妹細胞間のシナプス結合を解析することで、クラスター型プロトカドヘリンこそが姉妹細胞間に形成される双方向性シナプス結合の発達に必要な分子であることを明らかにしました。この発見は、姉妹神経細胞がお互いを認識する上で、クラスター型プロトカドヘリンが関与していることを世界で初めて証明したものです。
本研究で対象としたDnmt3bの遺伝子異常は、常染色体劣性遺伝病であるICF症候群の原因と考えられています。今回クラスター型プロトカドヘリンの遺伝子異常が生後のシナプス結合を障害する要因の一つと証明されたことにより、ICF症候群などさまざまな遺伝子異常を伴う疾患のメカニズムの解明と、治療法の開発に貢献できる可能性が示唆されます。
研究成果のポイント
- 大脳皮質の発生期に同じ神経幹細胞から生まれた姉妹神経細胞は、生後にお互いが選択的にシナプス結合を形成し合うことがわかりました。
- 胎生期の遺伝子発現制御を手がかりにして、将来的にシナプス結合が形成されることがわかりました。
- 生後の大脳皮質において姉妹神経細胞がお互いを認識する分子機構に細胞接着因子であるクラスター型プロトカドヘリンが関与することがわかりました。
本研究成果が社会に与える影響(本研究成果の意義)
脳の正常な発達には、あらかじめ遺伝的プログラムにより形成されたシナプス結合が、環境に適応できるように経験に伴い再調整されることが重要です。本研究成果は、遺伝子プログラミングにより形成されるシナプス結合の分子機構の一端を明らかにしたことに意義があります。(本研究で特徴づけたシナプス結合を元にして、経験や学習による神経結合の再調整が行われると予想されます。)また、本研究で対象とした分子に異常があると発達障害の原因になることが報告されているので、本成果がその病態メカニズムの理解、治療法の開発に役立つ可能性が期待されます。
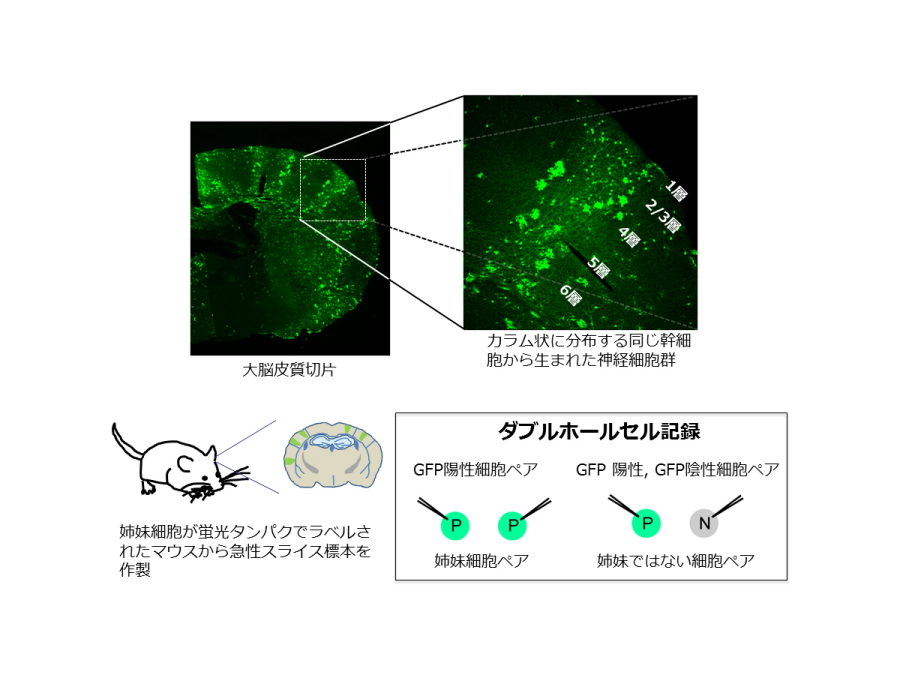
蛍光タンパクでラベルされた同じ幹細胞から生まれた神経細胞群と実験方法
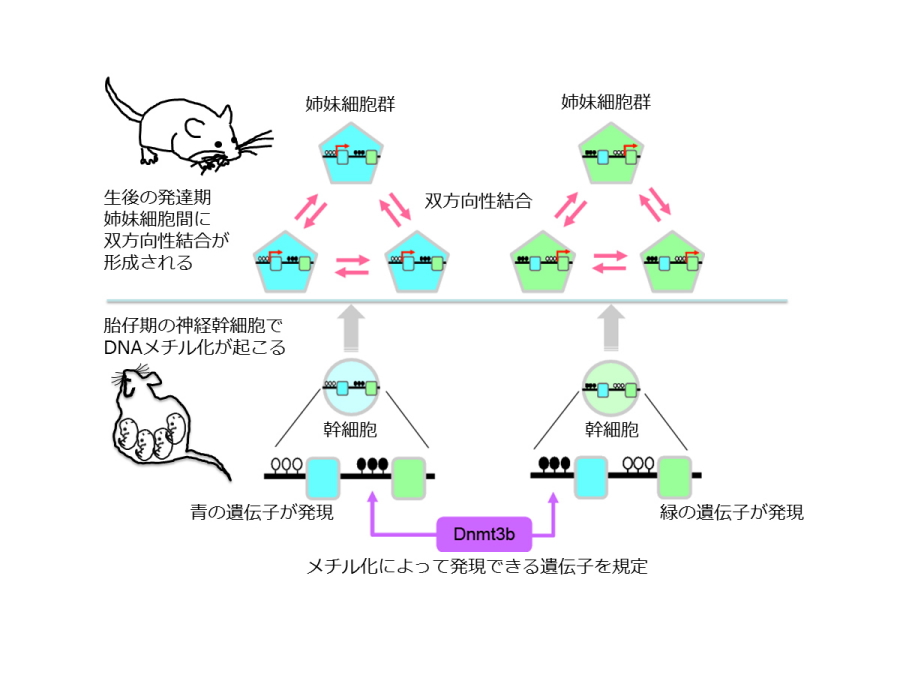
今回の発見内容の模式図
用語解説
- 神経幹細胞
神経細胞やグリア細胞を産生する細胞。脳のほとんどの領域においては、胎生期にのみ存在する。 - 姉妹神経細胞
大脳皮質の神経細胞は、胎生期に生まれる。神経細胞は、神経細胞を生み出す幹細胞から作られる。一つの幹細胞から生み出された神経細胞のことを姉妹細胞と呼ぶ。 - シナプス結合
神経細胞同士の接続部分で、神経細胞間の信号伝達が行われる結合。信号を送る側を前シナプスといい、信号を受け取る側を後シナプスと呼ぶ。このシナプスを介して細胞から細胞へ情報が伝えられる。 - 遺伝子プログラミング
生まれ持った遺伝子配列や遺伝子発現制御システムによってあらかじめプログラミングされた発生システム。 - 大脳皮質体性感覚野
大脳皮質の1領域で、触覚や痛覚の情報を受け取り、処理する領域。6層構造に分かれ、それぞれの層内、また異なる層の間で神経結合が形成されており、情報処理されると考えられている。本研究では、4層内において姉妹細胞間に双方向性結合が形成されやすいことを明らかにした。 - 双方向性シナプス結合
二つの神経細胞がお互いにシナプス結合すること。 - DNAメチル化酵素Dnmt3b
DNAの一部にメチル基を付加する酵素。これによりその遺伝子は発現できない状態になる。Dnmt3aとDnmt3bが存在し、発生初期にDnmt3bが発現し、発生後期Dnmt3bの減少とともにDnmt3aが発現する。 - クラスター型プロトカドヘリン
α、β、γに分けられる58種類の分子からなり、個々の神経細胞において、約15種類が発現する。その15種類の選択にDnmt3bによるDNAメチル化が関わることが知られている。細胞膜に発現し、同じ種類の分子同士は互いに接着する性質があり、細胞接着分子として知られる。 - ICF症候群
免疫力の低下、軽度の顔貌異常、染色体異常を特徴とする稀な常染色体劣性遺伝病。その原因遺伝子としてDnmt3bの異常が知られている。
| 原著論文 | BMC Biol 14, 103 (2016) |
|---|---|
| 論文タイトル | Establishment of high reciprocal connectivity between clonal cortical neurons is regulated by the Dnmt3b DNA methyltransferase and clustered protocadherins |
| 著者 | Tarusawa E (1, 2), Sanbo M (3), Okayama A (4), Miyashita T (1), Kitsukawa T (2, 4), Hirayama T (2, 4), Hirabayashi T (2, 4), Hasegawa S (2, 4), Kaneko R (5), Toyoda S (2, 4), Kobayashi T (6), Kato-Itoh M (6), Nakauchi H (6, 7), Hirabayashi M (2, 3, 8), Yagi T (2, 4), Yoshimura Y (1, 8)
|
| PubMed | 27912755 |
