ヒトでも!?受精卵にまで遡ってモニターできる新技術
細胞の系譜をたどり、疾患の発症メカニズムや生命現象の解明へ
| 原著論文 | Genome Res. (2022) |
|---|---|
| 論文タイトル | Early embryonic mutations reveal dynamics of somatic and germ cell lineages in mice |
| 研究室サイト | 心生物学研究室〈八木 健 教授〉 |
概要
放射線影響研究所分子生物科学部分子遺伝学研究室の内村有邦室長(大阪大学大学院生命機能研究科招へい教員)、大阪大学大学院生命機能研究科の八木健教授らの研究グループは、次世代シーケンサーを用いて、ゲノムDNA上で自然に発生する突然変異を調べることで、受精後の細胞分裂の過程をモニターすることが可能な新たな方法論の開発に成功しました(図1)。本方法を利用すれば、大人の組織サンプル(血液など)を解析するだけで、その個体が受精卵だったころまで遡り、受精後の細胞分裂の過程をモニターすることが可能になります(図2)。また、これにより、受精後に生じた個々の細胞系譜の追跡も可能になります。本方法は、遺伝子改変等を利用しないため、ヒトを対象とした解析にも利用可能だと考えられます。
本研究では、実験用マウスを用いた解析により、受精卵から、体細胞(体をつくる細胞)や生殖細胞(精子や卵子など)へと運命が決定されていくまでの細胞分裂の過程を再現することに成功しました(図3)。
細胞系譜は、個体発生や疾患の発症、老化の進行など、多くの生命現象に関わることが知られています。将来的には、新しい疾患治療方法の開発や病院での診断などに活用されていくことも期待されます。また、放射線や化学物質などで誘発される突然変異の研究や、生体へのリスク評価を行う上でも重要な技術になると考えられます。
研究の背景
精子と卵子が受精し、受精卵が形成されます。初めは1個の細胞である受精卵は、細胞分裂を繰り返し、2細胞、4細胞、8細胞へと増殖していき、ヒトの場合であれば、最終的には約37兆個の細胞に至り、ヒトひとりの体が構成されることになります。そのような発生過程における細胞分裂の由来(細胞系譜)を全て追跡することができれば、ヒトの体の成り立ちや各種疾患の発症メカニズム等をより深く理解していくことが可能になります。これまでの研究では、モデル動物にのみ利用可能な遺伝子組み換え技術等を利用した「細胞系譜の追跡技術」は知られていますが、ヒトを対象とした解析などにも簡単に利用できる「細胞系譜の追跡手法」は、これまで存在しませんでした。
本研究の成果
内村博士らの研究グループでは、次世代シーケンサーを用いて、体の中の一部の細胞だけに存在する突然変異(モザイク変異と呼ぶ)を正確に検出するための方法論を開発しました。また、「それらモザイク変異の生体中での存在頻度を正確に測定する方法」と「数理モデルを用いた独自の解析手法」を開発することで、大人の組織中に存在するモザイク変異を調べるだけで、その個体が受精した後の細胞分裂の過程を再現することが可能になりました(図2)。
実験用マウスを用いた解析では、マウス個体が、受精卵の時から、生殖細胞(精子や卵子のもとになる細胞)や体細胞(筋肉や神経細胞など、体を構成する細胞)へと細胞の運命決定がなされるまでの過程をモニターすることができました(図3)。また、受精後の細胞分裂では、細胞分裂あたり平均して1箇所の突然変異が新たに発生することも明らかにしました。これらの成果は、世界で初めての知見となります。
本方法を利用すれば、成体の組織サンプル(血液など)を利用するだけで、その個体の誕生(正確には受精)の瞬間に遡って、受精後の細胞分裂の過程をモニターすることが可能です。また、その際に生じた個々の細胞の系譜について受精卵から今にいたる経過を追跡することも可能になります。本方法は、発生生物学や様々な病気の原因や治療法を研究する上で重要な技術になると考えられます。
研究成果のポイント
- 次世代シーケンサーを用いて、成体の組織サンプル(血液など)を解析するだけで、受精卵だったころに遡って、受精後の細胞分裂の過程をモニターできる新技術の開発に成功
- この方法では、ゲノムDNA上で自然に発生する突然変異を利用して、受精後の細胞分裂の過程が時間の経過を経てもモニター可能
- ヒトの体は約37兆個の細胞から成る。今回の技術を利用すれば、各々の細胞が受精後の細胞分裂で生じたどの細胞の子孫にあたるか、その細胞の系譜を識別することが可能に
- 細胞系譜は、発生生物学や疾患の発症機構など、多くの生命現象に関わる。将来的には、新しい治療方法の開発や病院での診断などに活用されていくことも期待
- 開発した方法は、放射線や化学物質等で誘発される突然変異の研究にも利用可能
本研究成果が社会に与える影響(本研究成果の意義)
今回の研究はマウスを用いて行いましたが、同じ方法は、ヒトにも利用可能と考えられます。血液や口腔スワブなどのサンプルを採取し、そこに含まれるモザイク変異を解析すれば、被験者が母親の胎内で経験してきた受精後の1回目、2回目などの細胞分裂時に発生した突然変異の履歴が明らかになります。また、それらの突然変異を利用すれば、個々の細胞系譜の追跡も可能になります。
個々の細胞の系譜を捉えることは、様々な生命現象を理解していく上で特に重要です。例えば、個々の神経細胞の細胞系譜を調べることで、脳内における神経回路形成等の基盤原理の解明につながる可能性も考えられます。また、癌細胞の系譜を明らかにすることは、よりよい治療方法を選択するための重要な情報になる可能性があります。今回、開発に成功した手法は、今後、生命科学の幅広い分野で利用されていくことになると考えられます。将来的には、病院での診断や疾患リスクの推定などに利用されていく可能性も考えられます。
本方法は、突然変異の発生過程の解析にも利用可能であるため、放射線や化学物質等で誘発される突然変異の影響を解明するための方法論としても、広く利用されていくと考えられます。
研究者のコメント
人生で初めの細胞分裂である、受精後1回目の細胞分裂の時に誕生した細胞は、今の自分の体の中のどこにあるのだろうか?今回の研究では、そんな疑問に応えることができます。今生きている自分の体の一部を調べるだけで、自分の母親の胎内で行われた「受精後の様子」を復元できるようになります。将来的に研究が進めば、「受精後の様子」が分かるだけでなく、そこから自分自身の健康管理に役立つ情報が得られるようになると考えられます。(内村有邦)
特記事項
本研究成果は、2022年5月10日(火)午前2時(日本時間)に米国科学誌「Genome Research」(オンライン)に掲載されました。
なお、本研究は、大阪大学と放射線影響研究所が中心となり、日本学術振興会科学研究費補助金課題「胚発生期の自然発生変異に注目した高解像度な細胞系譜の解析」の一環として行われ、先進ゲノム支援(PAGS)による支援も受け、長崎大学情報データ科学部の松本拡高准教授、山梨大学発生工学研究センターの若山照彦教授、国立遺伝学研究所の豊田敦特任教授、東海大学医学部の権藤洋一教授らの協力を得て行われました。
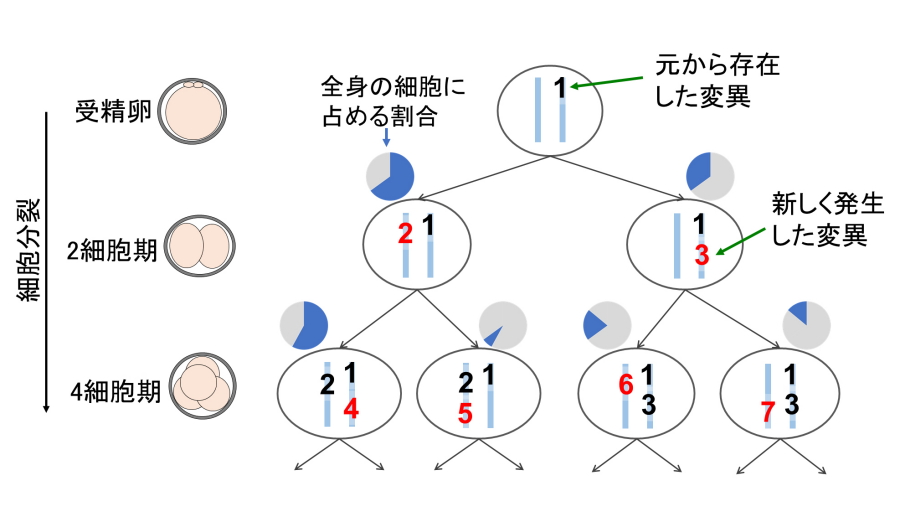
図1.突然変異を利用して受精後の細胞分裂の過程を再現
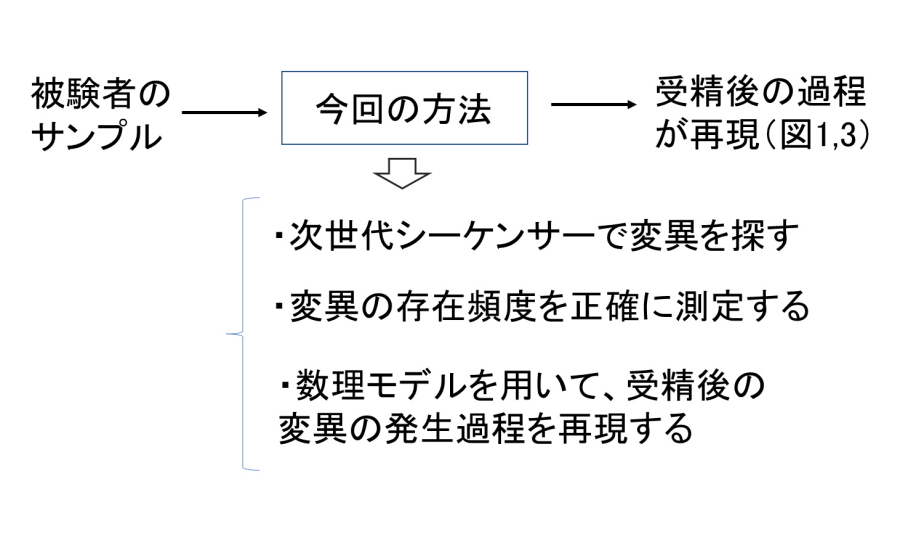
図2.研究方法の概要
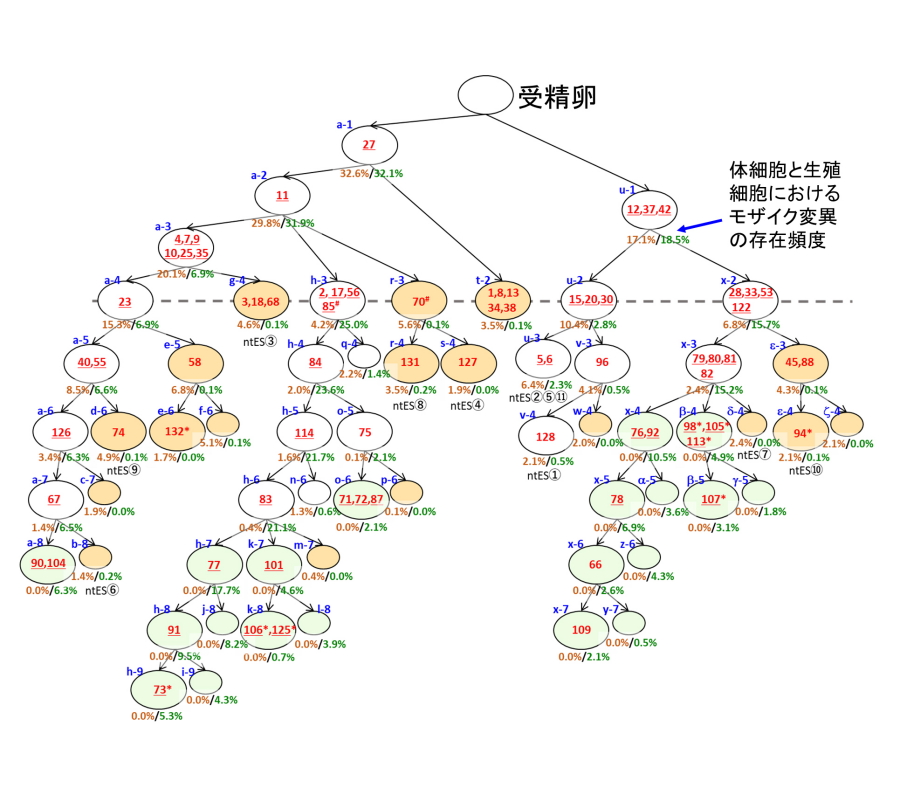
図3.マウスの実験で得られた実際の結果
赤数字は新たに発生した変異。緑色の細胞は生殖細胞(精子など)のみで、オレンジ色の細胞は体細胞の組織のみで観察された。
用語解説
- 次世代シーケンサー
ゲノムDNAの核酸の配列を調べるための装置。近年、技術的に大きな発展を遂げており、合計60億にも及ぶヒトやマウスのゲノムDNAの配列の解読も、現在は、実施可能である。 - ゲノムDNA
生物がもつ遺伝情報の実体。ヒトやマウスの場合であれば、4種類の核酸「A(アデニン)、T(チミン)、G(グアニン)、C(シトシン)」が、約30億個も連なる並び順(配列と呼ぶ)として構成されている。父親と母親の双方から1対(30億ずつ)を受け取ることで、ひとり分の遺伝情報となる。 - 突然変異
遺伝情報を担うゲノムDNAの並び順(配列)が変わることをDNAの突然変異と呼ぶ。細胞分裂を行うたびに突然変異は発生する。ヒトの場合、両親から子どもにゲノムDNAが伝播される際に、1世代あたり70~100か所程度の突然変異が発生することが知られている。
| 原著論文 | Genome Res. (2022) |
|---|---|
| 論文タイトル | Early embryonic mutations reveal dynamics of somatic and germ cell lineages in mice |
| 著者 | Arikuni Uchimura (1, 2), Hirotaka Matsumoto (3, 4), Yasunari Satoh (1), Yohei Minakuchi (5), Sayaka Wakayama (6), Teruhiko Wakayama (6, 7), Mayumi Higuchi (2), Masakazu Hashimoto (8), Ryutaro Fukumura (9), Atsushi Toyoda (5), Yoichi Gondo (9), Takeshi Yagi (2)
|
